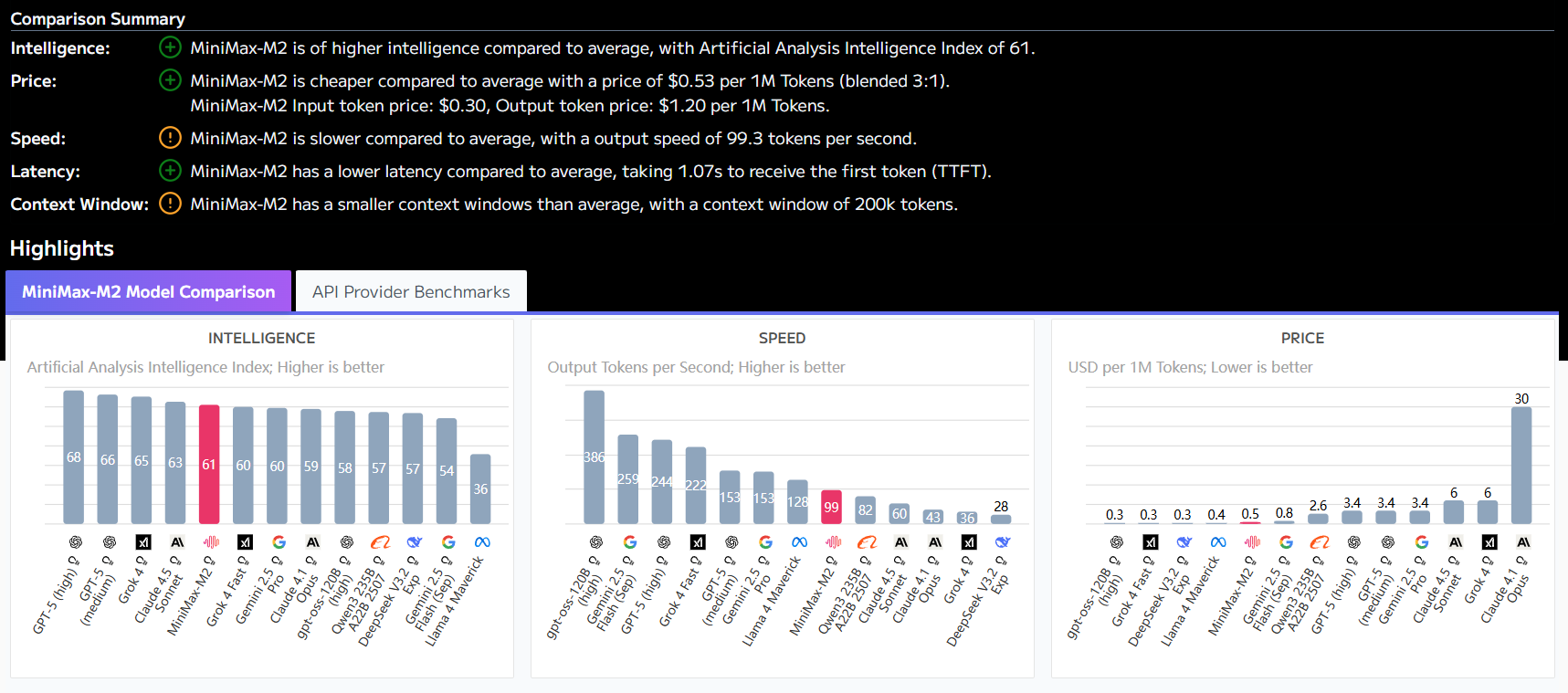
Rotas técnicas para migração de conhecimento entre domínios
Modelagem entre espécies usando o conjunto de dados OpenGenome2 do Evo 2:
- preparação básica::
- O download contém mais de 300 espécies
OpenGenome2.jsonlconjunto de dados - Instale a estrutura do Savanna:
pip install savanna-core
- O download contém mais de 300 espécies
- estratégia de ajuste fino::
- Congelar os parâmetros subjacentes:
freeze_layers: [0-25] - Ajuste das taxas de aprendizado:
lr=5e-5 - Configurar o cabeçalho de classificação de espécies:
cross_attention_heads: 8
- Congelar os parâmetros subjacentes:
- Métodos de validação::
- Use a validação cruzada k-fold (recomenda-se k=5)
- Calcule o índice de adaptação de domínio DAI > 0,85 para determinar o sucesso da migração
Aplicação típica: Ao migrar o conhecimento de BRCA1 treinado pelo genoma humano para um modelo de camundongo, recomenda-se uma estratégia de descongelamento progressivo (5 camadas por 10 épocas, começando pelo topo)
Essa resposta foi extraída do artigoEvo2: uma ferramenta de bioinformática de código aberto para apoiar a modelagem e o design de genomasO