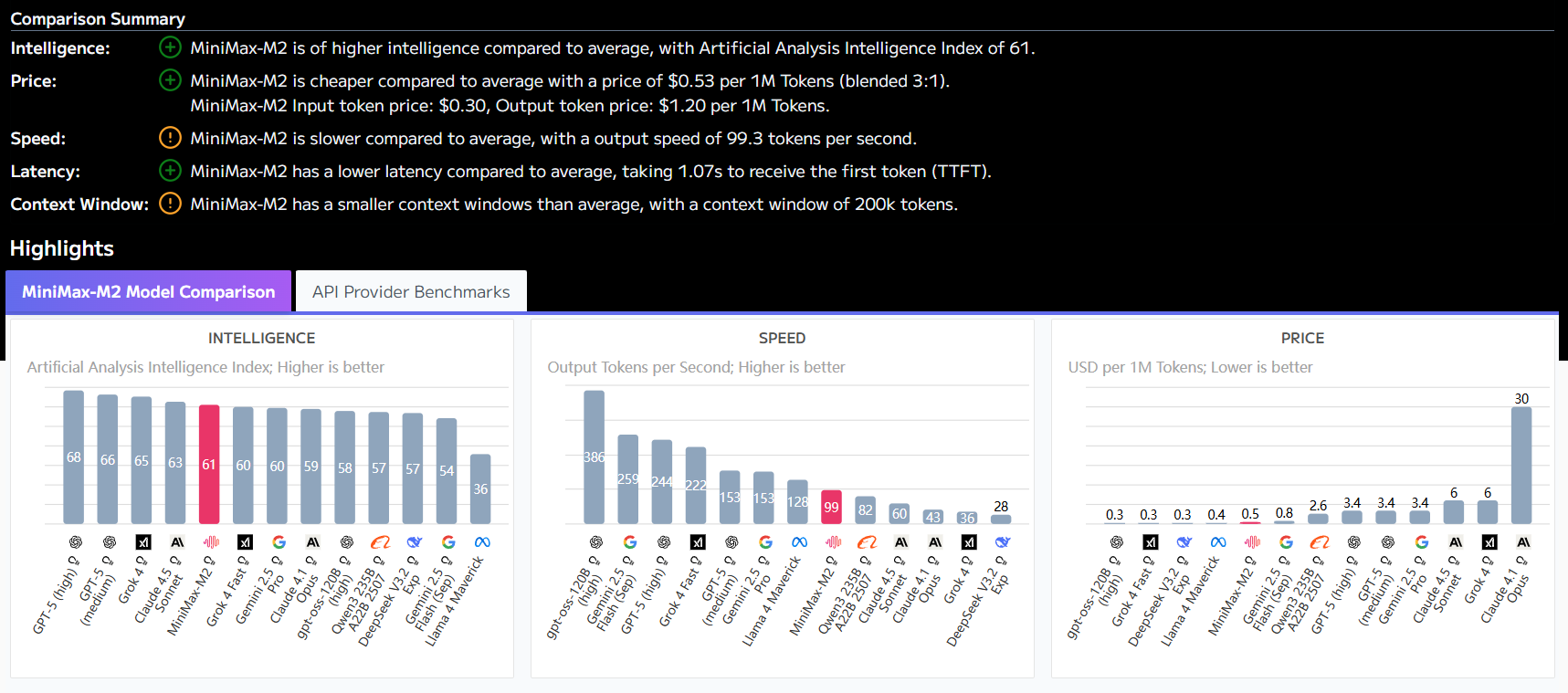
O recurso de previsão de efeito de variante de amostra zero do Evo 2 não requer treinamento adicional para avaliar os efeitos da variante do gene, e o processo de previsão do gene BRCA1 é o seguinte:
Etapas da previsão:
- Preparar dadosArmazenamento de sequências de referência e variantes em uma lista
ref_seqs = ["ATCG..."] # 参考序列
var_seqs = ["ATGG..."] # 变体序列 - Previsões operacionais::
ref_scores = model.score_sequences(ref_seqs)
var_scores = model.score_sequences(var_seqs)
print(f"Reference score: {ref_scores}, Variant score: {var_scores}")
Interpretação dos resultados:
A saída contém os valores de probabilidade das sequências de referência e variantes. Ao comparar as diferenças entre as duas:
- Pontuações mais baixas indicam que a variante pode ser prejudicial
- Pequenas alterações indicam variantes neutras
- Os escores aumentados podem ser uma variante de ganho de função
Essa função é particularmente aplicável à previsão da patogenicidade de genes relacionados ao câncer (por exemplo, BRCA1), fornecendo uma base de referência para a medicina de precisão.
Essa resposta foi extraída do artigoEvo2: uma ferramenta de bioinformática de código aberto para apoiar a modelagem e o design de genomasO