Por muito tempo, a tecnologia de IA médica de ponta ficou confinada atrás de licenças comerciais caras e sistemas opacos de "caixa preta". Isso tem desencorajado muitas organizações de pesquisa, equipes de desenvolvimento de pequeno e médio porte e médicos da linha de frente, retardando o processo de inovação tecnológica e adoção equitativa. Agora, uma nova iniciativa chamada OpenMed de um novo projeto está tentando resolver o impasse. O projeto foi lançado em Hugging Face lançou mais de 380 modelos avançados de reconhecimento de entidades nomeadas (NER) com base em textos médicos e clínicos e anunciou que, com base nos modelos de reconhecimento de entidades nomeadas (NER) Apache 2.0 A licença é gratuita e aberta para sempre.
A importância desse movimento não está apenas em oferecer uma alternativa gratuita, mas também no fato de que os modelos lançados superaram até mesmo as principais soluções comerciais de código fechado em vários benchmarks. Isso indica que as barreiras técnicas da IA na área de saúde estão sendo eliminadas pelo poder do código aberto.
Dilemas do setor e soluções de código aberto
O campo da IA na área da saúde enfrenta várias barreiras fundamentais para o crescimento:
- Altas taxas de licençaO alto custo do licenciamento das principais ferramentas comerciais de IA exclui as instituições acadêmicas e as startups com orçamentos limitados.
- Opacidade técnicaAs ferramentas comerciais normalmente não divulgam a arquitetura do modelo, os dados de treinamento e o funcionamento, o que dificulta a avaliação da confiabilidade e da possível parcialidade pelos usuários.
- Lenta iteração de tecnologiaAlguns dos modelos pagos não conseguiram acompanhar os últimos avanços da tecnologia de IA, e seu desempenho ficou gradualmente aquém das pesquisas de ponta no meio acadêmico.
- aplicação limitadaA IA de alta qualidade está concentrada nas mãos de algumas grandes empresas, o que limita a inclusão da tecnologia.
OpenMed O projeto responde diretamente a esses desafios. Ele oferece mais de 380 cursos gratuitos NER modelos que se concentram na identificação de entidades-chave em textos médicos, como nomes de medicamentos, doenças, genes, estruturas anatômicas etc. Esses modelos possuem os seguintes recursos importantes:
- ✅ Totalmente gratuitoBaseado em
Apache 2.0Licença que permite o uso, a modificação e a distribuição gratuitos. - ✅ pronto para usoProjetado para cenários do mundo real, ele pode ser implementado sem muito trabalho extra.
- ✅ Flexibilidade de tamanhoO número de parâmetros do modelo varia de 109M a 568M para atender a diferentes requisitos de hardware.
- ✅ comprovadoTestes rigorosos de desempenho em mais de 13 conjuntos de dados padrão na área médica.
- ✅ ecologicamente compatívelObservação: Em conjunto com o
Hugging Face和PyTorchIntegração perfeita com as principais estruturas, como
OpenMed Detalhes do kit de ferramentas
OpenMed A biblioteca de modelos foi cuidadosamente ajustada e testada na Gellus Pontuações F1 em conjuntos de dados como 0.998demonstrando seu desempenho superior.
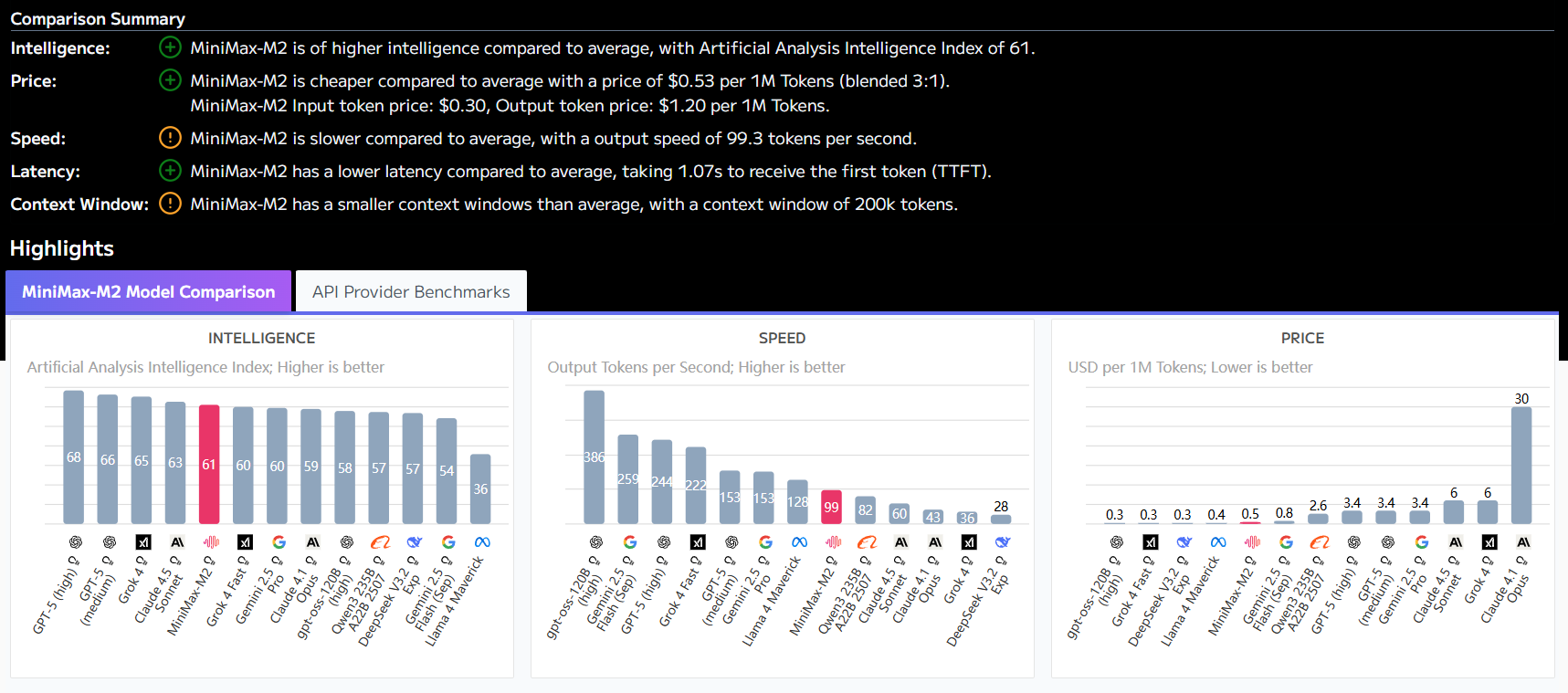
Comparação de desempenho: código aberto OpenMed e modelos de negócios de código fechado
Para visualizar sua competitividade, aOpenMed publicou uma comparação de desempenho com os atuais modelos de negócios de código fechado de última geração. Os dados mostram queOpenMed Ele não só é comparável aos modelos comerciais em vários conjuntos de dados, como também alcança um desempenho significativamente superior em alguns cenários.
| conjunto de dados | Melhor pontuação F1 da OpenMed (%) | Fração SOTA F1 de fonte fechada (%)† | Gap (OpenMed - SOTA) | atual líder em código fechado |
|---|---|---|---|---|
| BC4CHEMD | 95.40 | 94.39 | +1.01 | Spark NLP BertForTokenClassification |
| BC5CDR-Chem | 96.10 | 94.88 | +1.22 | Spark NLP BertForTokenClassification |
| BC5CDR-Disease | 91.20 | 88.5 | +2.70 | BioMegatron |
| NCBI-Disease | 91.10 | 89.71 | +1.39 | BioBERT |
| JNLPBA | 81.90 | 82.00 | –0.10 | KeBioLM (knowledge-enhanced LM) |
| Linnaeus | 96.50 | 92.70 | +3.80 | BERN2 toolkit |
| Species-800 | 86.40 | 82.59 | +3.81 | Spark NLP BertForTokenClassification |
| BC2GM | 90.10 | 88.75 | +1.35 | Spark NLP Bi-LSTM-CNN-Char |
| AnatEM | 90.60 | 91.65 | –1.05 | Spark NLP BertForTokenClassification |
| BioNLP 2013 CG | 89.90 | 87.83 | +2.07 | Spark NLP BertForTokenClassification |
| Gellus | 99.80 | 63.40 | +36.40 | ConNER |
| CLL | 95.70 | 85.98 | — | (Nenhum SOTA emitido) |
| FSU | 96.10 | — | — | (Nenhum SOTA emitido) |
† As pontuações de código fechado são derivadas dos melhores resultados publicados, revisados por pares ou classificados na literatura (geralmente modelos comerciais, como Spark NLP, NEEDLE, BERN2, etc.).
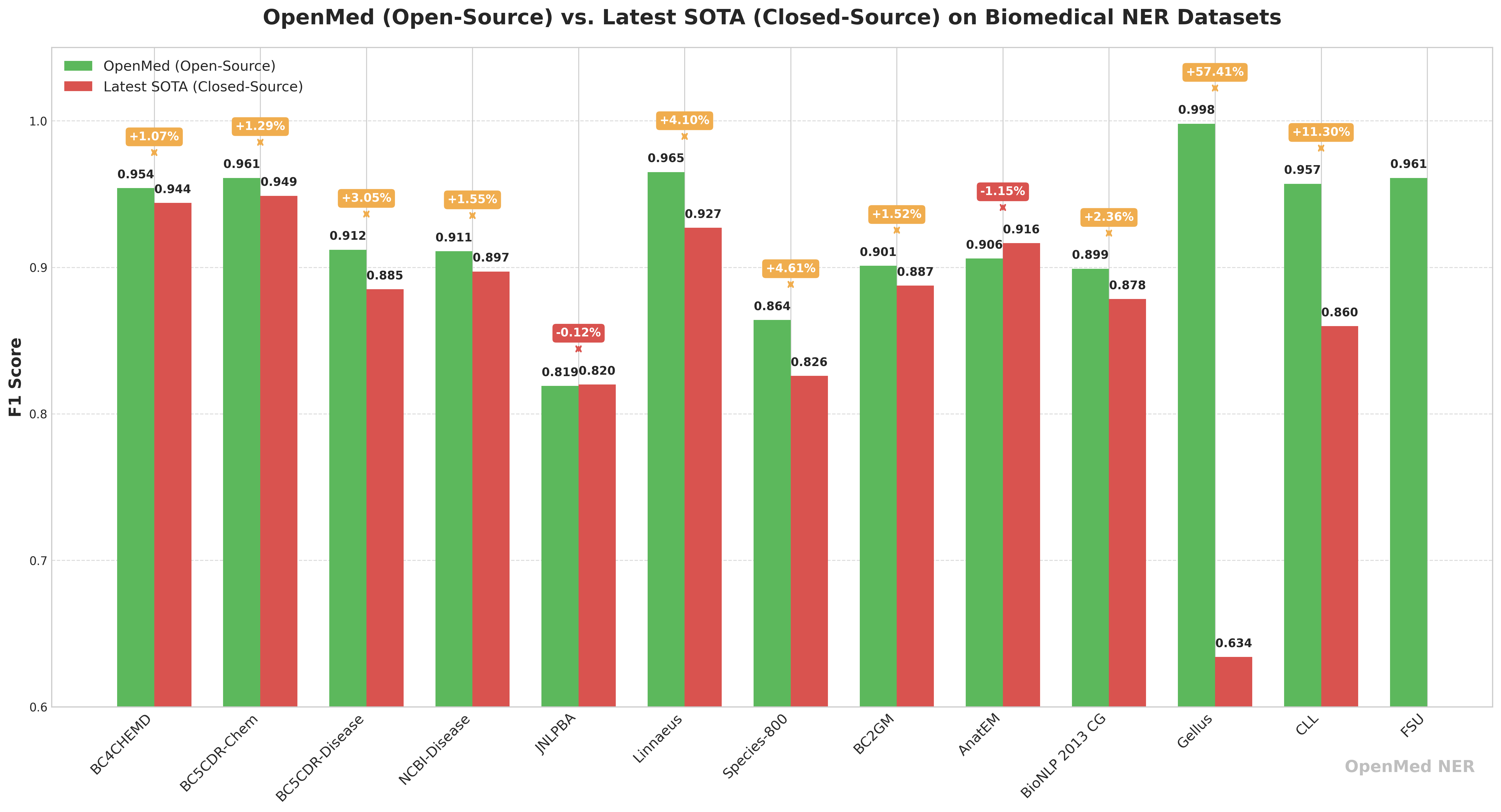
Particularmente preocupante é o fato de que em Gellus no conjunto de dados.OpenMed A pontuação F1 de 36,41 do TP3T é maior do que a do melhor modelo anterior, o que sugere que os modelos de código aberto voltados para a otimização têm grande potencial em tarefas específicas.
Por área de aplicação
A tabela abaixo mapeia os conjuntos de dados para seus domínios de saúde correspondentes e recomenda os modelos correspondentes com base no desempenho combinado dos conjuntos de dados em cada domínio.
| reino | Conjuntos de dados incluídos | Número de modelos disponíveis | Faixa de parâmetros | Modelos de recomendação |
|---|---|---|---|---|
| farmacologia | bc5cdr_chem, bc4chemd, fsu |
90 | 109M – 568M | OpenMed-NER-PharmaDetect-SuperClinical-434M |
| Doença/Patologia | bc5cdr_disease, ncbi_disease |
60 | 109M – 434M | OpenMed-NER-PathologyDetect-PubMed-v2-109M |
| genômica | jnlpba, bc2gm, species800, linnaeus, gellus |
150 | 335M – 568M | OpenMed-NER-GenomicDetect-SnowMed-568M |
| anatomia | anatomy |
30 | 560M | OpenMed-NER-AnatomyDetect-ElectraMed-560M |
| estudo de tumores | bionlp2013_cg |
30 | 355M | OpenMed-NER-OncologyDetect-SuperMedical-355M |
| registro clínico | cll |
30 | 560M | OpenMed-NER-BloodCancerDetect-ElectraMed-560M |
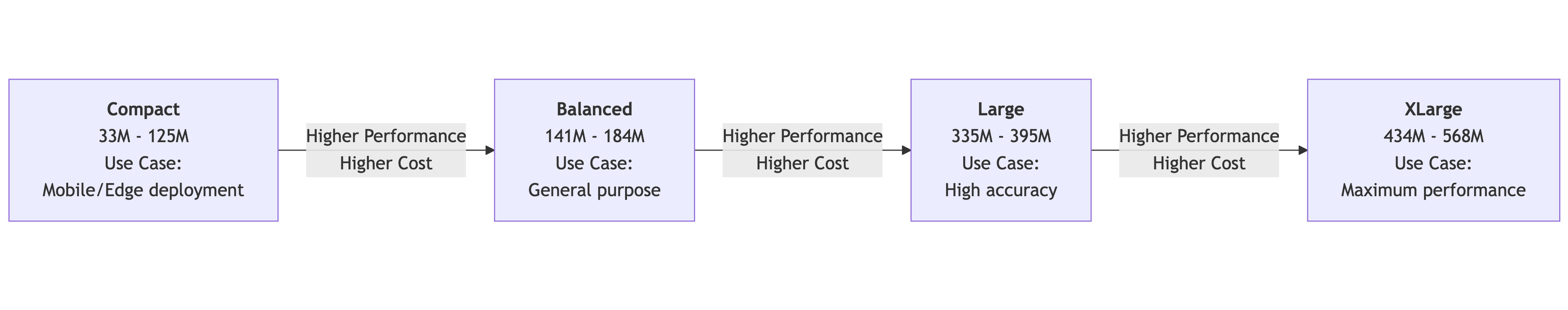
⚡️ Selecione por tamanho do modelo
| magnitude | quantidade de participantes | Cenários ideais |
|---|---|---|
| compacto | 109M | Prototipagem rápida e ambiente com poucos recursos |
| mega | 335M – 355M | Escolha equilibrada de precisão e desempenho |
| mega | 434M | Profissionais versáteis com excelente desempenho |
| gigante | 560M – 568M | Uma missão de extrema precisão |
O melhor modelo em cada conjunto de dados
Abaixo está um resumo dos modelos com melhor desempenho em cada conjunto de dados e suas pontuações e tamanhos de F1.
| conjunto de dados | melhor modelo | Pontuação da F1 | Tamanho do modelo (parâmetro) |
|---|---|---|---|
bc5cdr_chem |
OpenMed-NER-PharmaDetect-SuperClinical-434M |
0.961 | 434M |
bionlp2013_cg |
OpenMed-NER-OncologyDetect-SuperMedical-355M |
0.899 | 355M |
bc4chemd |
OpenMed-NER-ChemicalDetect-PubMed-335M |
0.954 | 335M |
linnaeus |
OpenMed-NER-SpeciesDetect-PubMed-335M |
0.965 | 335M |
jnlpba |
OpenMed-NER-DNADetect-SuperClinical-434M |
0.819 | 434M |
bc5cdr_disease |
OpenMed-NER-DiseaseDetect-SuperClinical-434M |
0.912 | 434M |
fsu |
OpenMed-NER-ProteinDetect-SnowMed-568M |
0.961 | 568M |
ncbi_disease |
OpenMed-NER-PathologyDetect-PubMed-v2-109M |
0.911 | 109M |
bc2gm |
OpenMed-NER-GenomeDetect-SuperClinical-434M |
0.901 | 434M |
cll |
OpenMed-NER-BloodCancerDetect-ElectraMed-560M |
0.957 | 560M |
gellus |
OpenMed-NER-GenomicDetect-SnowMed-568M |
0.998 | 568M |
anatomy |
OpenMed-NER-AnatomyDetect-ElectraMed-560M |
0.906 | 560M |
species800 |
OpenMed-NER-OrganismDetect-BioMed-335M |
0.864 | 335M |
Começando rapidamente e aumentando a escala
alavancagem Hugging Face Transformers Biblioteca, integração OpenMed O processo de modelagem é muito simples e pode ser invocado em apenas três linhas de código.
from transformers import pipeline
ner_pipeline = pipeline("token-classification", model="OpenMed/OpenMed-NER-PharmaDetect-SuperClinical-434M", aggregation_strategy="simple")
text = "Patient prescribed 10mg aspirin for hypertension."
entities = ner_pipeline(text)
print(entities)
# 输出: [{'entity_group': 'CHEMICAL', 'score': 0.99..., 'word': 'aspirin', 'start': 28, 'end': 35}]
Para cenários em que conjuntos de dados em grande escala precisam ser processados, o projeto também oferece soluções eficientes de processamento em lote.
from transformers.pipelines.pt_utils import KeyDataset
from datasets import Dataset, load_dataset
import pandas as pd
# 加载公开的医疗数据集(使用一个子集进行测试)
medical_dataset = load_dataset("BI55/MedText", split="train[:100]")
data = pd.DataFrame({"text": medical_dataset["Completion"]})
dataset = Dataset.from_pandas(data)
# 使用适合您硬件的批处理大小
batch_size = 16 # 根据您的 GPU 显存进行调整
results = []
ner_pipeline = pipeline("token-classification", model="OpenMed/OpenMed-NER-PharmaDetect-SuperClinical-434M", device=0) # 使用GPU
for out in ner_pipeline(KeyDataset(dataset, "text"), batch_size=batch_size):
results.extend(out)
print(f"批处理完成 {len(results)} 条文本")
NER O valor da tecnologia desbloqueada no mundo real
A tecnologia de Reconhecimento de Entidades Nomeadas (NER) é capaz de extrair e classificar automaticamente informações importantes de textos não estruturados. Na área da saúde, essa tecnologia é um catalisador para ativar o valor de grandes quantidades de dados em anotações clínicas, registros de pacientes e literatura científica.
- Proteção da privacidade do paciente (desidentificação de dados):
NERAs informações pessoais de saúde (PHI), como nome, endereço etc., podem ser automaticamente identificadas e removidas dos registros médicos. Isso é importante para proteger a privacidade do paciente, cumprir comHIPAAe outras leis e regulamentações, ao mesmo tempo em que fornece uma fonte de dados compatível e segura para pesquisas médicas que é muito mais eficiente e precisa do que o processamento manual. - Construção de gráficos de conhecimento médico (extração de relacionamento de entidades)Depois de identificar entidades, como medicamentos, doenças etc., outras técnicas podem analisar as relações entre elas (por exemplo, "O medicamento A causa o efeito colateral B"). Isso ajuda a criar um gráfico de conhecimento médico que dá suporte à tomada de decisões clínicas, acelera o desenvolvimento de medicamentos e, por fim, possibilita o tratamento personalizado.
- 💡 Otimização dos custos e do gerenciamento do atendimento médico (códigos HCC)A codificação da Categoria de Condição Hierárquica (HCC) é um processo fundamental usado pelos pagadores do setor de saúde, como o Medicare, para projetar custos e definir taxas de reembolso.
NERAs informações de diagnóstico podem ser extraídas automaticamente dos registros médicos para auxiliar na codificação, garantindo que os provedores sejam remunerados de forma justa pelo tratamento de casos complexos e ajudando a identificar pacientes de alto risco para intervenção proativa.
Ao promover a automação dessas tarefas essenciais, aNER A tecnologia está transformando textos médicos inativos em soluções acionáveis que aumentam a segurança dos dados, aceleram a pesquisa, melhoram o prognóstico dos pacientes e reduzem os custos operacionais.OpenMed O surgimento de um novo sistema, sem dúvida, acelerará consideravelmente o processo.