Recentemente, a OpenAI e a startup de biotecnologia Retro Biosciences anunciaram os resultados de uma colaboração que demonstra o enorme potencial da inteligência artificial nas ciências da vida. Usando um modelo de IA, o GPT-4b micro, projetado especificamente para a engenharia de proteínas, a equipe conseguiu aumentar em mais de 50 vezes a eficiência da expressão dos principais marcadores para a reprogramação induzida de células-tronco.
O núcleo dessa pesquisa concentra-se nos fatores Yamanaka, um grupo de proteínas que ganhou o Prêmio Nobel por seu papel pioneiro na reprogramação celular. Essas proteínas podem transformar células adultas diferenciadas, como as células da pele, em "células-tronco pluripotentes induzidas" (iPSCs) com o potencial de se desenvolver em praticamente qualquer tecido. Essa capacidade abre novos caminhos para o tratamento de cegueira, diabetes, infertilidade e até mesmo para solucionar a escassez de órgãos.
No entanto, a aplicação dos fatores Yamanaka tradicionais é extremamente ineficiente, com normalmente menos de 0,1% de células sendo transformadas com sucesso e todo o processo levando mais de três semanas. A eficiência cai ainda mais, especialmente quando se trata de células de doadores idosos ou doentes. Desta vez, a variante da proteína reprojetada pela IA não apenas melhorou significativamente a eficiência, mas também demonstrou maior capacidade de reparo de danos ao DNA, o que significa que ela tem maior potencial para o rejuvenescimento celular.
Essa descoberta inicial, feita em 2025, agora foi validada por experimentos replicados em uma ampla gama de doadores, tipos de células e métodos de entrega, confirmando a pluripotência completa e a estabilidade genômica das linhas de células iPSC resultantes.
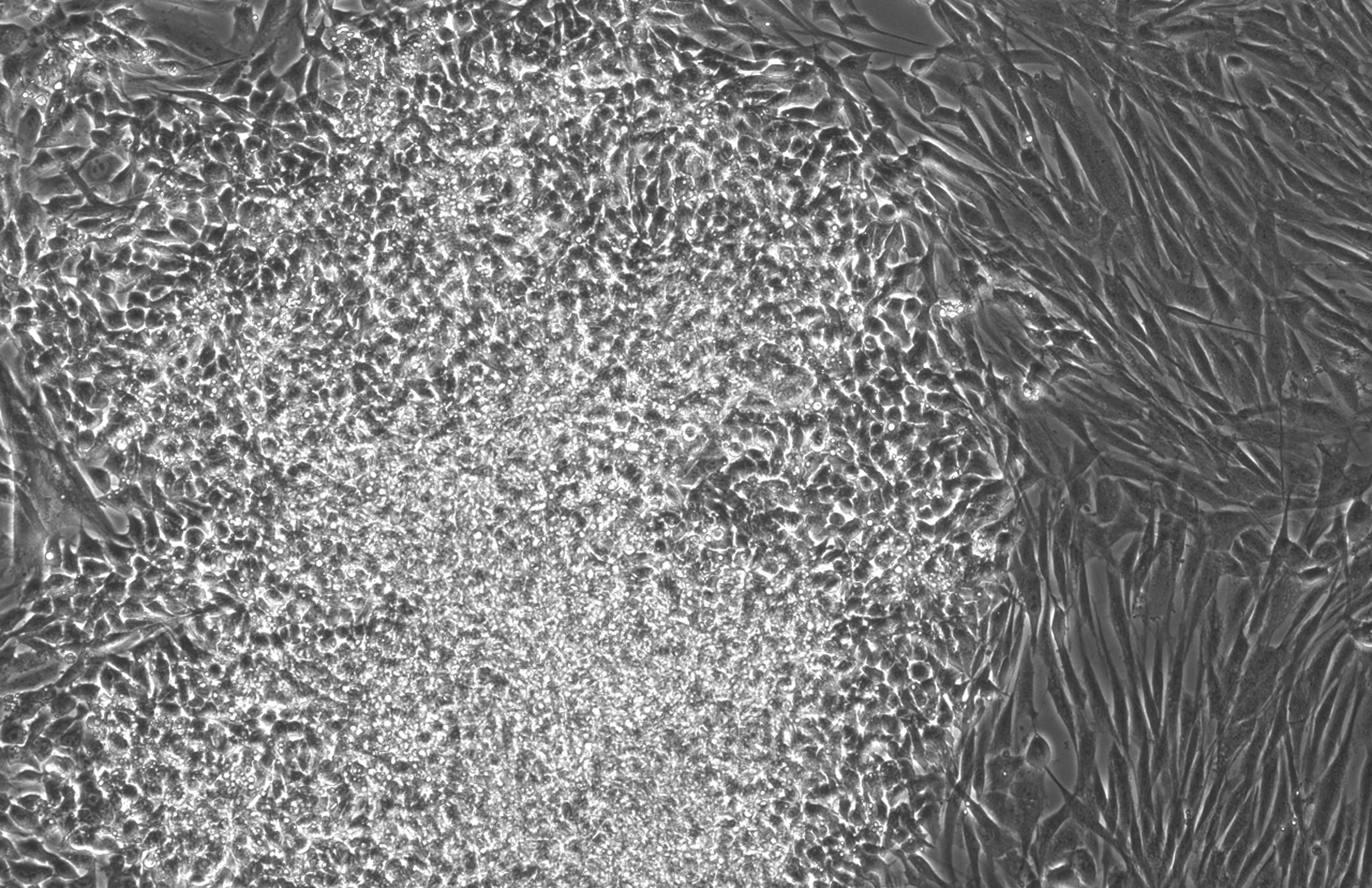
Modelos experimentais de GPT adaptados para engenharia de proteínas
Para validar que a IA pode acelerar a pesquisa em ciências biológicas, a OpenAI criou um modelo personalizado chamado GPT-4b micro. O modelo é uma versão em miniatura do GPT-4o que foi especialmente treinado para proporcionar a ele um profundo conhecimento de biologia, especialmente nas áreas de engenharia de proteínas para controle e flexibilidade.
Diferentemente da maioria dos modelos de linguagem de proteínas, os dados de treinamento do micro GPT-4b contêm não apenas sequências de proteínas, mas também uma combinação de texto biológico e dados estruturais em 3D rotulados. Os dados de treinamento são particularmente enriquecidos com informações contextuais, como descrições textuais de proteínas, sequências homólogas co-evoluídas e proteomas de interação conhecidos. Essa abordagem permite que o modelo gere sequências com base em dicas de atributos específicos e lide igualmente bem com proteínas estruturadas e proteínas "intrinsecamente desordenadas". Os fatores Yamanaka são os últimos, e sua atividade depende de um grande número de interações transitórias com vários parceiros de ligação, em vez de uma única estrutura estável fixa.
Dessa forma, o comprimento efetivo do contexto do modelo excede em muito o limite de sequências independentes, e até 64.000 podem ser processadas no processo de inferência token o que não tem precedentes na modelagem de sequências de proteínas.
Transformação assistida por inteligência artificial de SOX2 e KLF4
O fator Yamanaka é composto por quatro proteínas: OCT4, SOX2, KLF4 e MYC (OSKM). Otimizá-las diretamente por meio da modificação das sequências de proteínas é uma tarefa difícil. No caso da SOX2 (que contém 317 aminoácidos) e da KLF4 (que contém 513 aminoácidos), por exemplo, o número de variantes possíveis é de até 10 elevado à milésima potência.
Os métodos tradicionais de "evolução dirigida", em que apenas alguns resíduos de aminoácidos são alterados por vez, podem explorar apenas uma pequena fração das possibilidades. A IA, por outro lado, pode explorar um espaço de design muito mais amplo. A equipe da Retro Biosciences primeiro montou uma plataforma de triagem em laboratório úmido e depois usou o micro GPT-4b para gerar uma série de sequências candidatas denominadas "RetroSOX".
Os resultados foram surpreendentes: na triagem, as sequências sugeridas pelo modelo que excederam 30% superaram o SOX2 do tipo selvagem na expressão dos principais marcadores de pluripotência, embora suas sequências de aminoácidos diferissem em mais de 100, em média. Em contraste, nas telas convencionais, os acertos eram geralmente inferiores a 10%.
Em seguida, a equipe visou o KLF4. O modelo gerou 14 variantes "RetroKLF" que superaram a melhor combinação na tela RetroSOX, com uma taxa de acerto próxima a 50%.
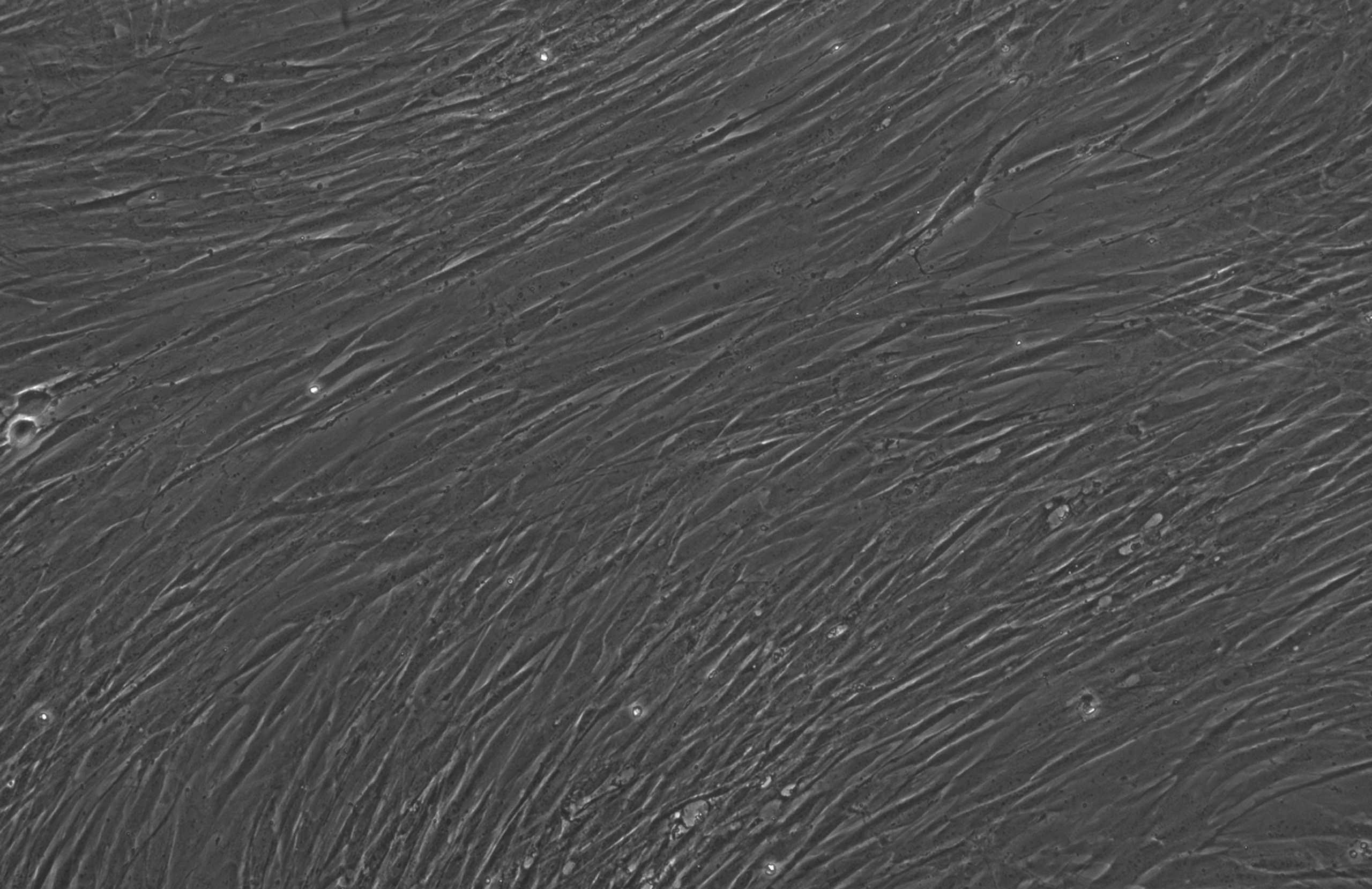
Os efeitos foram mais dramáticos quando as variantes RetroSOX e RetroKLF superiores foram combinadas. Em três experimentos independentes, os fibroblastos apresentaram um aumento significativo nos marcadores de pluripotência precoce e tardia, e os marcadores tardios apareceram vários dias antes do que com a mistura OSKM do tipo selvagem. Outros testes, como a coloração com fosfatase alcalina (AP), também confirmaram que essas colônias de células não apenas expressavam marcadores tardios, mas também apresentavam forte atividade de AP, um forte indicador de pluripotência.
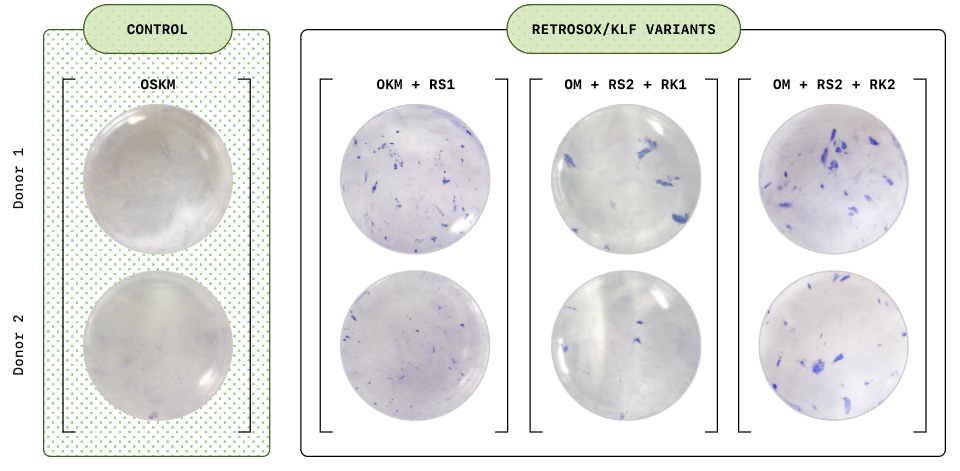
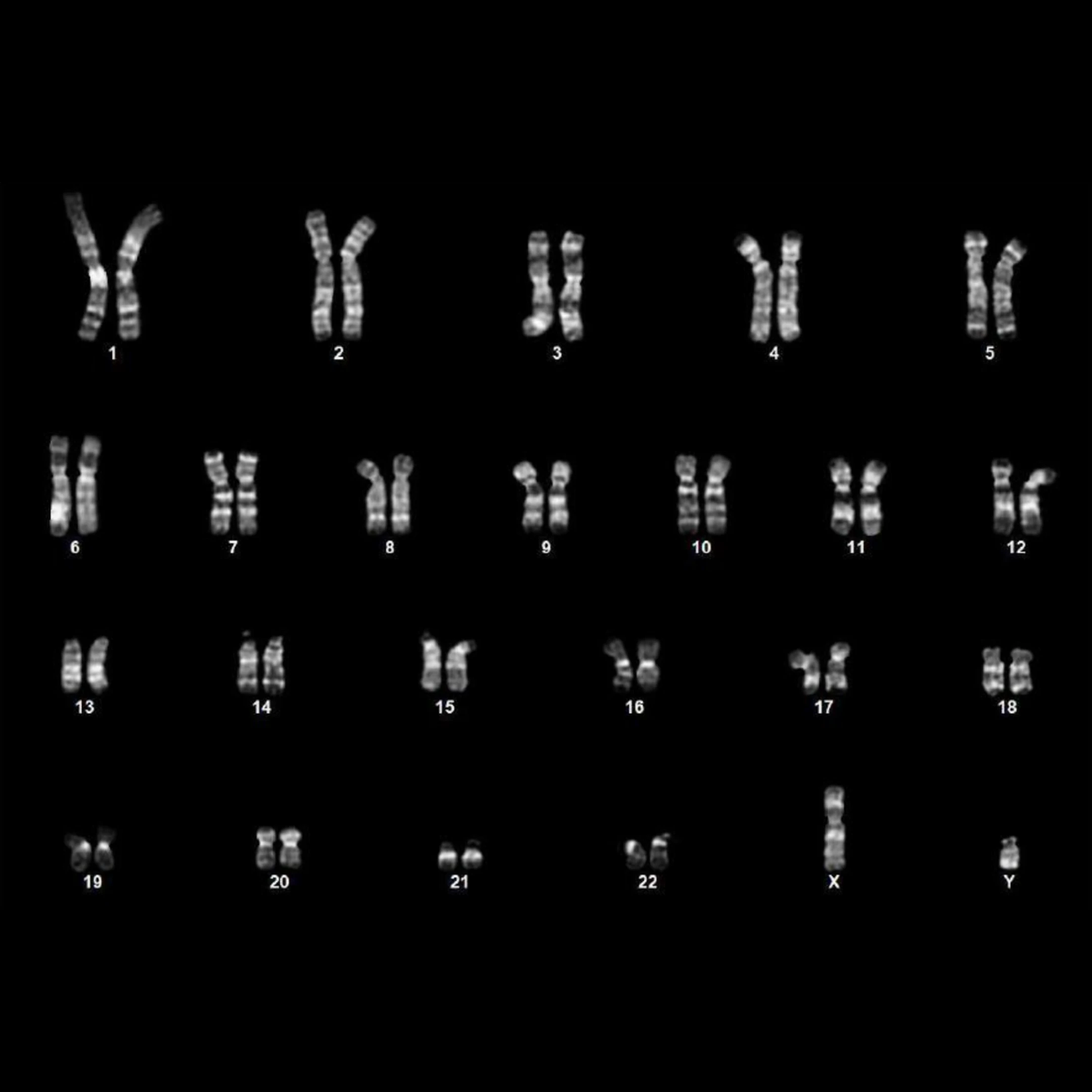
Para explorar o potencial clínico, a equipe também testou um método de entrega diferente (mRNA alternativo aos vetores virais) e outro tipo de célula - células estromais mesenquimais (MSCs) de três doadores de meia-idade com mais de 50 anos. Em apenas 7 dias, mais de 301 célulasTP3T começaram a expressar os principais marcadores de pluripotência; no 12º dia, mais de 851 célulasTP3T ativaram marcadores endógenos de células-tronco, incluindo OCT4 e NANOG. A cariotipagem dessas células mostrou uma estrutura cromossômica normal, confirmando sua estabilidade genômica e adequação à terapia celular.
Reparo aprimorado de danos ao DNA
Além de melhorar a eficiência da reprogramação, os pesquisadores exploraram o potencial dessas variantes projetadas para o rejuvenescimento celular, especialmente a capacidade de reparar danos ao DNA, uma das características clássicas da senescência celular.
Na análise de danos ao DNA, após o tratamento com produtos químicos genotóxicos, as células que expressam a mistura RetroSOX/KLF apresentaram marcadores de quebra de fita dupla de DNA (sinais γ-H2AX) significativamente menores do que as células que usam OSKM padrão ou controles. Isso sugere que as variantes de proteína projetadas pela IA podem reparar danos ao DNA com mais eficiência, fornecendo um novo caminho possível para retardar a senescência celular.

perspectivas futuras
Esse trabalho demonstra claramente a rapidez com que um modelo de IA específico de um domínio pode alcançar avanços em problemas científicos específicos. Quando os pesquisadores combinam percepções profundas do domínio com ferramentas de modelagem de linguagem, problemas que antes levavam anos para serem resolvidos agora podem progredir em dias.
É claro que essa pesquisa ainda está em um estágio inicial, e a segurança e os efeitos de longo prazo ainda precisam ser cuidadosamente avaliados antes de passar do laboratório para a aplicação clínica. Mas, sem dúvida, ela abre uma nova porta para a aplicação de IA no campo da biomedicina, anunciando a chegada de uma nova era de medicina personalizada e medicina regenerativa orientada por IA.