长期以来,尖端的医疗人工智能技术一直被禁锢在高昂的商业许可和不透明的“黑箱”系统之后。这使得许多研究机构、中小型开发团队和一线医生望而却步,减缓了技术创新和公平应用的进程。如今,一个名为 OpenMed 的新项目正试图打破这一僵局。该项目在 Hugging Face 社区发布了超过380个先进的医疗与临床文本命名实体识别(NER)模型,并宣布基于 Apache 2.0 许可证永久免费开放。
此举的意义不仅在于提供免费的替代品,更在于其发布的模型在多个基准测试中,性能甚至超越了主流的商业闭源方案。这预示着医疗AI领域的技术壁垒正在被开源力量瓦解。
行业困境与开源方案
医疗AI领域的发展面临着几个核心障碍:
- 高昂的许可费用:顶级的商业AI工具许可费用高昂,将预算有限的学术机构和初创公司排除在外。
- 技术不透明:商业工具通常不公开其模型架构、训练数据和工作原理,用户难以评估其可靠性和潜在偏见。
- 技术迭代缓慢:部分付费模型未能跟上最新的AI技术进展,性能逐渐落后于学术界的前沿研究。
- 应用受限:优质的AI能力集中在少数大型企业手中,限制了技术普惠。
OpenMed 项目直接回应了这些挑战。它提供的 380 多个免费 NER 模型,专注于识别医学文本中的关键实体,如药物名称、疾病、基因、解剖结构等。这些模型具备以下突出特点:
- ✅ 完全免费:基于
Apache 2.0许可证,允许自由使用、修改和分发。 - ✅ 即用性:专为实际应用场景设计,无需大量额外工作即可部署。
- ✅ 尺寸灵活:模型参数量从 109M 到 568M 不等,适应不同硬件需求。
- ✅ 经过验证:在超过 13 个医疗领域的标准数据集上进行了严格的性能测试。
- ✅ 生态兼容:与
Hugging Face和PyTorch等主流框架无缝集成。
OpenMed 工具箱详解
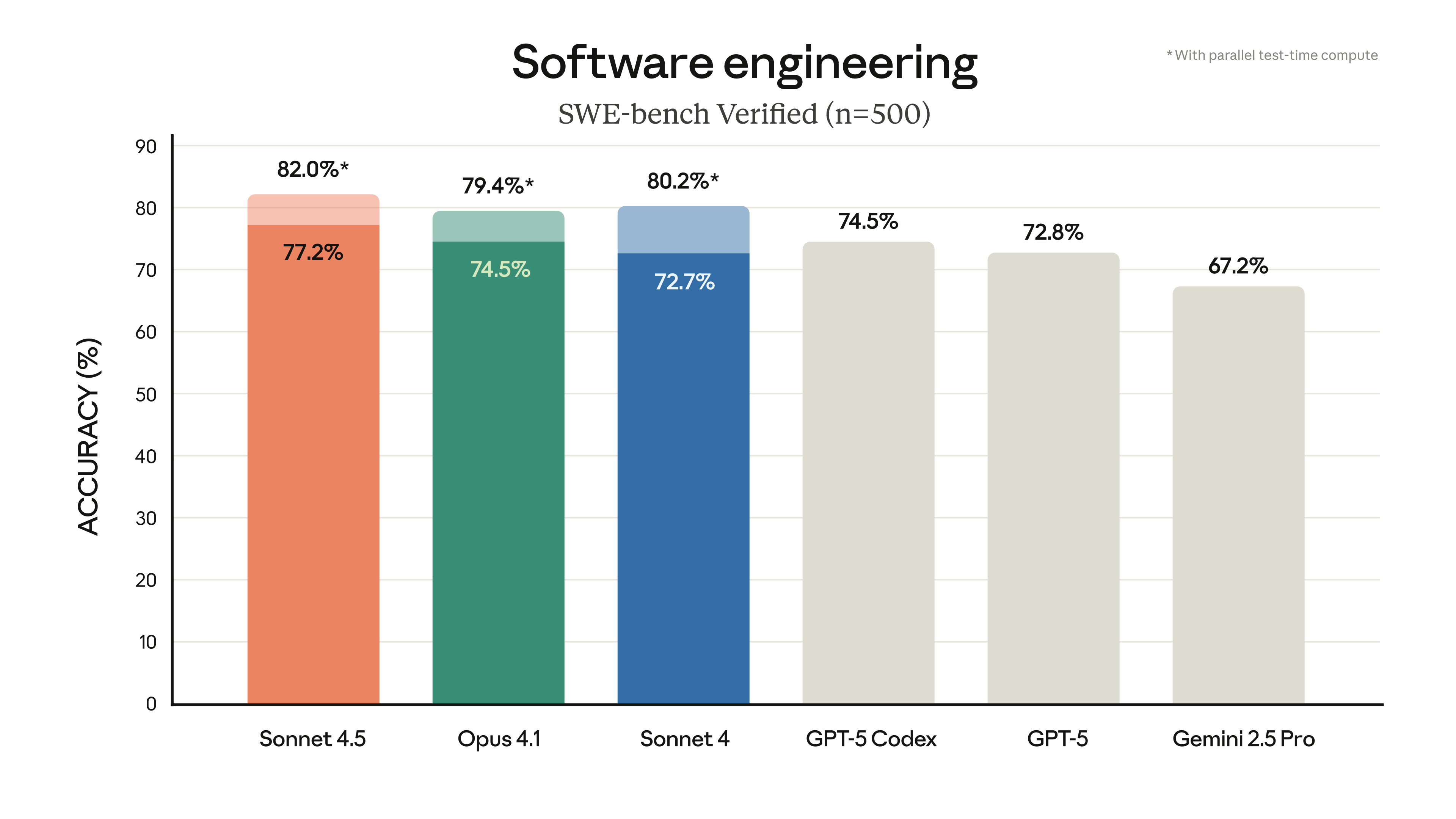
OpenMed 的模型库经过精心微调和测试,在 Gellus 等数据集上的 F1 分数高达 0.998,展示了其卓越的性能。
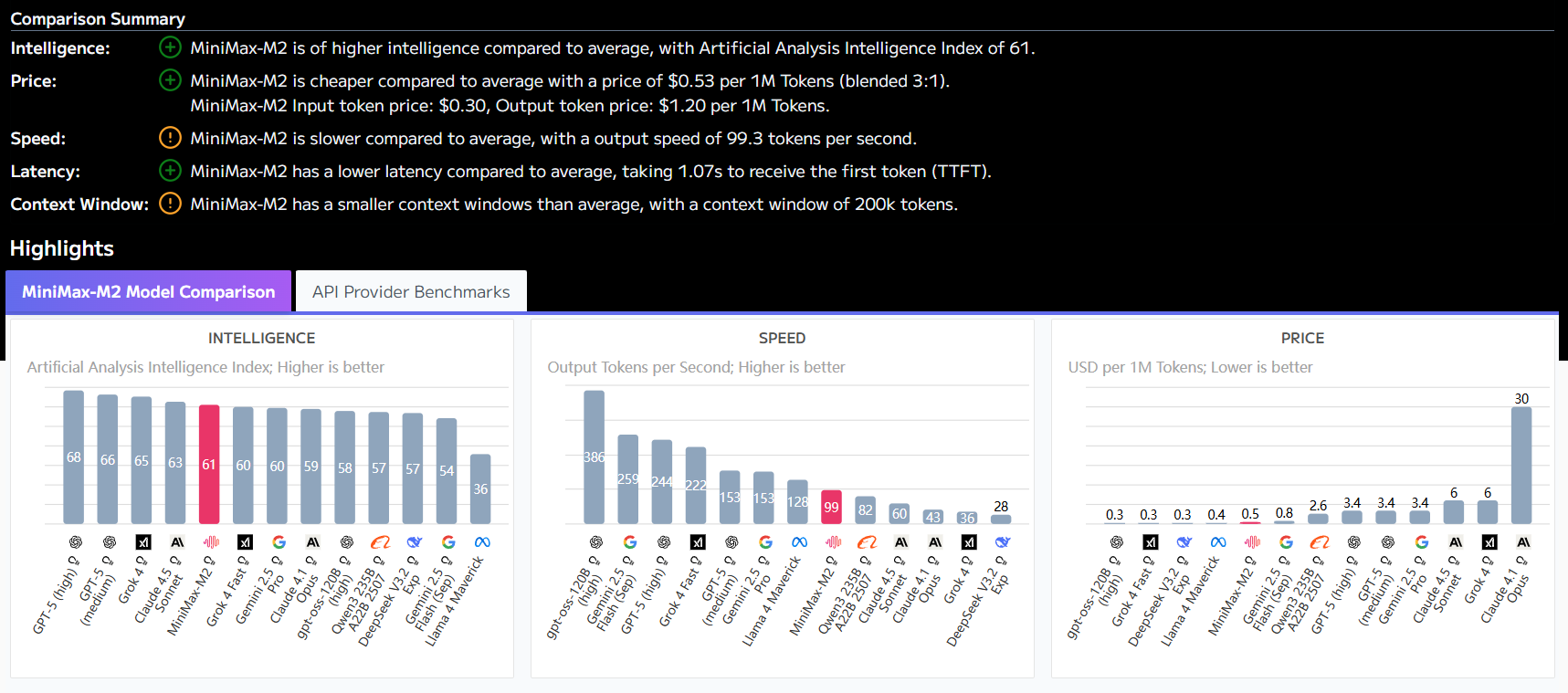
🔬 性能对比:开源 OpenMed 与闭源商业模型
为了直观展示其竞争力,OpenMed 公布了与当前最先进的闭源商业模型的性能对比。数据显示,OpenMed 不仅在多个数据集上与商业模型相当,更在某些场景下实现了显著超越。
| 数据集 | OpenMed 最佳 F1 分数 (%) | 闭源 SOTA F1 分数 (%)† | 差距 (OpenMed – SOTA) | 当前闭源领先者 |
|---|---|---|---|---|
| BC4CHEMD | 95.40 | 94.39 | +1.01 | Spark NLP BertForTokenClassification |
| BC5CDR-Chem | 96.10 | 94.88 | +1.22 | Spark NLP BertForTokenClassification |
| BC5CDR-Disease | 91.20 | 88.5 | +2.70 | BioMegatron |
| NCBI-Disease | 91.10 | 89.71 | +1.39 | BioBERT |
| JNLPBA | 81.90 | 82.00 | –0.10 | KeBioLM (knowledge-enhanced LM) |
| Linnaeus | 96.50 | 92.70 | +3.80 | BERN2 toolkit |
| Species-800 | 86.40 | 82.59 | +3.81 | Spark NLP BertForTokenClassification |
| BC2GM | 90.10 | 88.75 | +1.35 | Spark NLP Bi-LSTM-CNN-Char |
| AnatEM | 90.60 | 91.65 | –1.05 | Spark NLP BertForTokenClassification |
| BioNLP 2013 CG | 89.90 | 87.83 | +2.07 | Spark NLP BertForTokenClassification |
| Gellus | 99.80 | 63.40 | +36.40 | ConNER |
| CLL | 95.70 | 85.98 | — | (无已发布的 SOTA) |
| FSU | 96.10 | — | — | (无已发布的 SOTA) |
† 闭源分数来源于文献中已发表的最高同行评审或排行榜结果(通常是 Spark NLP、NEEDLE、BERN2 等商业模型)。
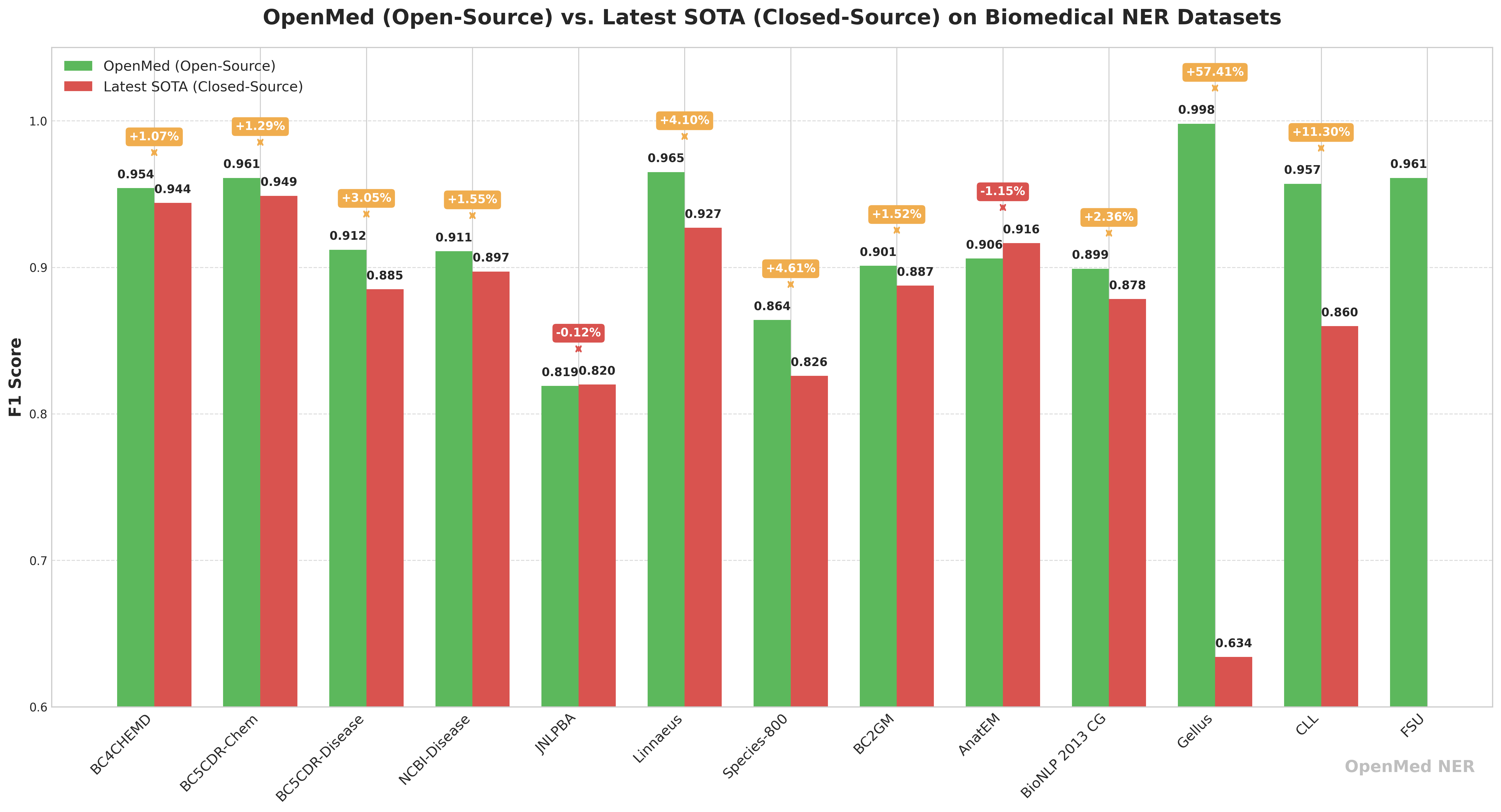
尤其值得关注的是,在 Gellus 数据集上,OpenMed 的 F1 分数高出先前最佳模型 36.4%,这表明在特定任务上,专注优化的开源模型拥有巨大的潜力。
🔬 按应用领域划分
下表将数据集与其对应的医疗领域进行映射,并根据各领域数据集的综合表现推荐了相应的模型。
| 领域 | 包含的数据集 | 可用模型数量 | 参数量范围 | 推荐模型 |
|---|---|---|---|---|
| 药理学 | bc5cdr_chem, bc4chemd, fsu |
90 个 | 109M – 568M | OpenMed-NER-PharmaDetect-SuperClinical-434M |
| 疾病/病理学 | bc5cdr_disease, ncbi_disease |
60 个 | 109M – 434M | OpenMed-NER-PathologyDetect-PubMed-v2-109M |
| 基因组学 | jnlpba, bc2gm, species800, linnaeus, gellus |
150 个 | 335M – 568M | OpenMed-NER-GenomicDetect-SnowMed-568M |
| 解剖学 | anatomy |
30 个 | 560M | OpenMed-NER-AnatomyDetect-ElectraMed-560M |
| 肿瘤学 | bionlp2013_cg |
30 个 | 355M | OpenMed-NER-OncologyDetect-SuperMedical-355M |
| 临床记录 | cll |
30 个 | 560M | OpenMed-NER-BloodCancerDetect-ElectraMed-560M |
⚡️ 按模型大小选择
| 大小 | 参数量 | 最佳适用场景 |
|---|---|---|
| 紧凑型 | 109M | 快速原型和低资源环境 |
| 大型 | 335M – 355M | 精度与性能的均衡选择 |
| 超大型 | 434M | 全能型,性能优异 |
| 巨大型 | 560M – 568M | 追求极致精度的任务 |
📊 各数据集上的最佳模型
以下是每个数据集上表现最佳的模型及其 F1 分数和大小的摘要。
| 数据集 | 最佳模型 | F1 分数 | 模型大小 (参数) |
|---|---|---|---|
bc5cdr_chem |
OpenMed-NER-PharmaDetect-SuperClinical-434M |
0.961 | 434M |
bionlp2013_cg |
OpenMed-NER-OncologyDetect-SuperMedical-355M |
0.899 | 355M |
bc4chemd |
OpenMed-NER-ChemicalDetect-PubMed-335M |
0.954 | 335M |
linnaeus |
OpenMed-NER-SpeciesDetect-PubMed-335M |
0.965 | 335M |
jnlpba |
OpenMed-NER-DNADetect-SuperClinical-434M |
0.819 | 434M |
bc5cdr_disease |
OpenMed-NER-DiseaseDetect-SuperClinical-434M |
0.912 | 434M |
fsu |
OpenMed-NER-ProteinDetect-SnowMed-568M |
0.961 | 568M |
ncbi_disease |
OpenMed-NER-PathologyDetect-PubMed-v2-109M |
0.911 | 109M |
bc2gm |
OpenMed-NER-GenomeDetect-SuperClinical-434M |
0.901 | 434M |
cll |
OpenMed-NER-BloodCancerDetect-ElectraMed-560M |
0.957 | 560M |
gellus |
OpenMed-NER-GenomicDetect-SnowMed-568M |
0.998 | 568M |
anatomy |
OpenMed-NER-AnatomyDetect-ElectraMed-560M |
0.906 | 560M |
species800 |
OpenMed-NER-OrganismDetect-BioMed-335M |
0.864 | 335M |
快速上手与规模化应用
借助 Hugging Face Transformers 库,集成 OpenMed 模型的过程非常简单,只需三行代码即可调用。
from transformers import pipeline
ner_pipeline = pipeline("token-classification", model="OpenMed/OpenMed-NER-PharmaDetect-SuperClinical-434M", aggregation_strategy="simple")
text = "Patient prescribed 10mg aspirin for hypertension."
entities = ner_pipeline(text)
print(entities)
# 输出: [{'entity_group': 'CHEMICAL', 'score': 0.99..., 'word': 'aspirin', 'start': 28, 'end': 35}]
对于需要处理大规模数据集的场景,该项目也提供了高效的批处理方案。
from transformers.pipelines.pt_utils import KeyDataset
from datasets import Dataset, load_dataset
import pandas as pd
# 加载公开的医疗数据集(使用一个子集进行测试)
medical_dataset = load_dataset("BI55/MedText", split="train[:100]")
data = pd.DataFrame({"text": medical_dataset["Completion"]})
dataset = Dataset.from_pandas(data)
# 使用适合您硬件的批处理大小
batch_size = 16 # 根据您的 GPU 显存进行调整
results = []
ner_pipeline = pipeline("token-classification", model="OpenMed/OpenMed-NER-PharmaDetect-SuperClinical-434M", device=0) # 使用GPU
for out in ner_pipeline(KeyDataset(dataset, "text"), batch_size=batch_size):
results.extend(out)
print(f"批处理完成 {len(results)} 条文本")
NER 技术解锁的真实世界价值
命名实体识别(NER)技术能够从非结构化的文本中自动提取和分类关键信息。在医疗领域,这项技术是激活临床笔记、患者记录和科研文献中海量数据价值的催化剂。
- 🔒 患者隐私保护(数据去标识化):
NER可以自动识别并移除医疗记录中的个人健康信息(PHI),如姓名、地址等。这在保护患者隐私、遵守HIPAA等法律法规的同时,为医学研究提供了合规、安全的数据来源,其效率和准确性远超手动处理。 - 🔗 医疗知识图谱构建(实体关系提取):在识别出药物、疾病等实体后,进一步的技术可以分析它们之间的关系(如“药物 A 导致副作用 B”)。这有助于构建医疗知识图谱,为临床决策提供支持,加速药物研发,并最终实现个性化治疗。
- 💡 优化医疗成本与管理(HCC 编码):分级病情类别(HCC)编码是医疗支付方(如 Medicare)用于预测成本和设定报销率的关键流程。
NER可从病历中自动提取诊断信息以辅助编码,确保医疗机构因治疗复杂病例获得公平报酬,同时帮助识别高风险患者以进行主动干预。
通过推动这些关键任务的自动化,NER 技术正在将沉睡的医疗文本转化为可操作的解决方案,从而提升数据安全性、加速研究进程、改善患者预后并降低运营成本。OpenMed 的出现,无疑将极大地加速这一进程。