Viel zu lange war die medizinische KI-Spitzentechnologie hinter kostspieligen kommerziellen Lizenzen und undurchsichtigen "Blackbox"-Systemen verborgen. Dies hat viele Forschungseinrichtungen, kleine und mittlere Entwicklungsteams und Ärzte an vorderster Front abgeschreckt und den Prozess der technologischen Innovation und der gerechten Übernahme verlangsamt. Jetzt wird eine neue Initiative mit der Bezeichnung OpenMed eines neuen Projekts versucht, den Stillstand zu überwinden. Das Projekt wurde gestartet in Hugging Face Community hat über 380 fortschrittliche medizinische und klinische textbasierte Modelle zur Erkennung von benannten Entitäten (NER) veröffentlicht und angekündigt, dass auf der Grundlage der Apache 2.0 Die Lizenz ist kostenlos und zeitlich unbegrenzt gültig.
Die Bedeutung dieses Schrittes liegt nicht nur in der Bereitstellung einer kostenlosen Alternative, sondern auch in der Tatsache, dass die veröffentlichten Modelle in mehreren Benchmarks sogar die kommerziellen Closed-Source-Lösungen der Mainstream-Unternehmen übertrafen. Dies ist ein Zeichen dafür, dass die technischen Barrieren im Bereich der KI im Gesundheitswesen durch die Kraft von Open Source abgebaut werden.
Dilemmas der Industrie und Open-Source-Lösungen
Im Bereich der KI im Gesundheitswesen gibt es mehrere Haupthindernisse für das Wachstum:
- Hohe LizenzgebührenDie hohen Kosten für die Lizenzierung kommerzieller KI-Tools schließen akademische Einrichtungen und Start-ups mit begrenzten Budgets aus.
- Technische OpazitätKommerzielle Tools legen ihre Modellarchitektur, ihre Trainingsdaten und ihre Arbeitsweise in der Regel nicht offen, so dass es für die Benutzer schwierig ist, ihre Zuverlässigkeit und mögliche Verzerrungen zu beurteilen.
- Langsame Technologie-IterationEinige der kostenpflichtigen Modelle haben nicht mit den neuesten Fortschritten in der KI-Technologie Schritt gehalten, und ihre Leistung ist allmählich hinter der Spitzenforschung in der Wissenschaft zurückgeblieben.
- begrenzte AnwendungHochwertige KI-Fähigkeiten sind in den Händen einiger weniger Großunternehmen konzentriert, was die Integration der Technologie einschränkt.
OpenMed Das Projekt ist eine direkte Antwort auf diese Herausforderungen. Es bietet mehr als 380 kostenlose NER Modelle, die sich auf die Identifizierung von Schlüsselentitäten in medizinischen Texten konzentrieren, wie z. B. Namen von Medikamenten, Krankheiten, Genen, anatomischen Strukturen, usw. Diese Modelle weisen die folgenden herausragenden Merkmale auf:
- ✅ Völlig kostenlos: Basierend auf
Apache 2.0Die Lizenz erlaubt die freie Nutzung, Veränderung und Verbreitung. - ✅ gebrauchsfertigeEntwickelt für reale Szenarien, kann es ohne großen Mehraufwand implementiert werden.
- ✅ Flexibilität bei der GrößeDie Anzahl der Modellparameter reicht von 109M bis 568M, um unterschiedlichen Hardwareanforderungen gerecht zu werden.
- ✅ bewährtStrenge Leistungstests mit über 13 Standarddatensätzen aus dem medizinischen Bereich.
- ✅ ökologisch verträglich:: In Verbindung mit dem
Hugging Face和PyTorchNahtlose Integration in gängige Frameworks wie
OpenMed Details zum Toolkit
OpenMed Die Modellbibliothek wurde sorgfältig aufeinander abgestimmt und in der Gellus F1-Bewertungen für Datensätze wie 0.998und beweist damit seine überlegene Leistung.
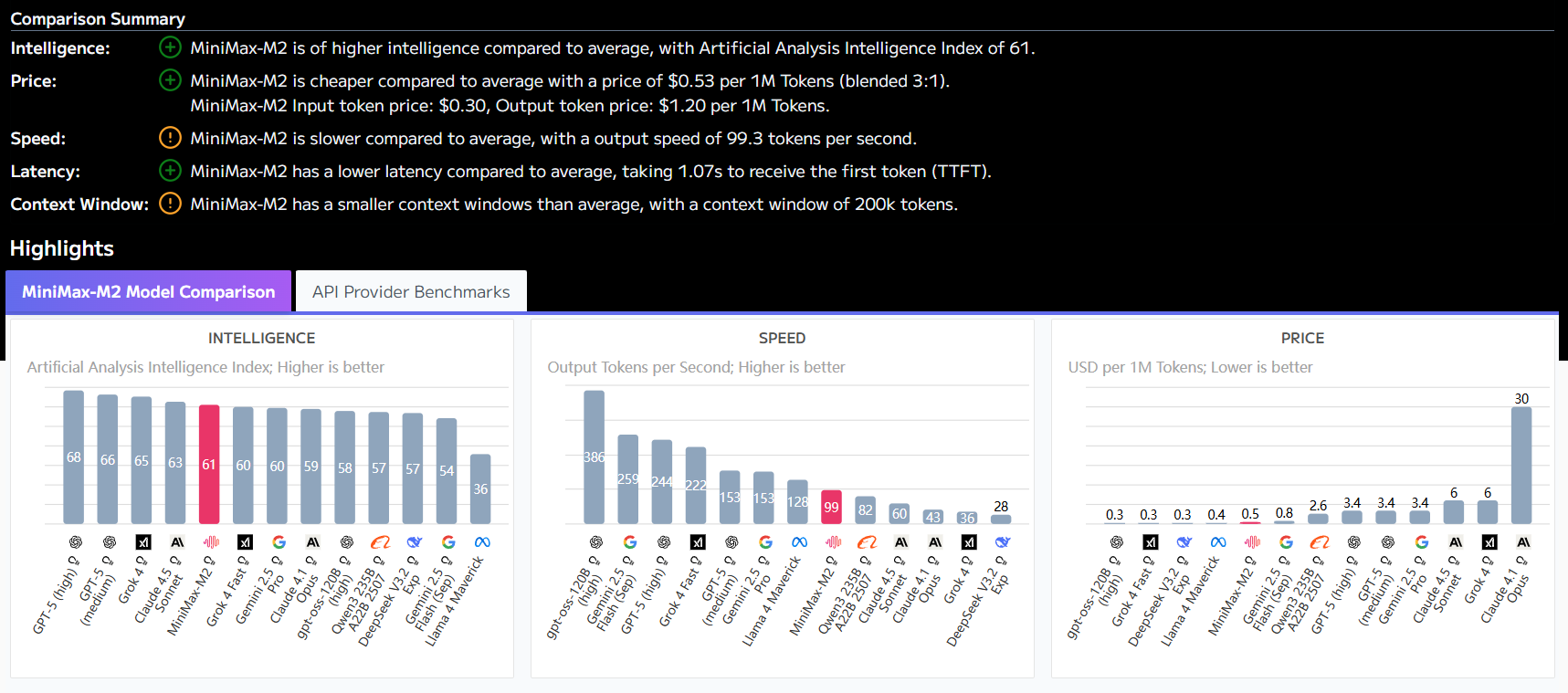
🔬 Leistungsvergleich: Open Source OpenMed und Closed-Source-Geschäftsmodelle
Um seine Wettbewerbsfähigkeit zu veranschaulichen, hat dieOpenMed veröffentlichte einen Leistungsvergleich mit den derzeit modernsten Closed-Source-Geschäftsmodellen. Die Daten zeigen, dassOpenMed Es ist nicht nur mit kommerziellen Modellen vergleichbar, sondern übertrifft sie in einigen Szenarien deutlich.
| Datensatz | OpenMed Best F1 Score (%) | Geschlossene Quelle SOTA F1 Fraktion (%)† | Lücke (OpenMed - SOTA) | derzeitiger Marktführer im Bereich Closed-Source |
|---|---|---|---|---|
| BC4CHEMD | 95.40 | 94.39 | +1.01 | Spark NLP BertForTokenClassification |
| BC5CDR-Chem | 96.10 | 94.88 | +1.22 | Spark NLP BertForTokenClassification |
| BC5CDR-Disease | 91.20 | 88.5 | +2.70 | BioMegatron |
| NCBI-Disease | 91.10 | 89.71 | +1.39 | BioBERT |
| JNLPBA | 81.90 | 82.00 | –0.10 | KeBioLM (knowledge-enhanced LM) |
| Linnaeus | 96.50 | 92.70 | +3.80 | BERN2 toolkit |
| Species-800 | 86.40 | 82.59 | +3.81 | Spark NLP BertForTokenClassification |
| BC2GM | 90.10 | 88.75 | +1.35 | Spark NLP Bi-LSTM-CNN-Char |
| AnatEM | 90.60 | 91.65 | –1.05 | Spark NLP BertForTokenClassification |
| BioNLP 2013 CG | 89.90 | 87.83 | +2.07 | Spark NLP BertForTokenClassification |
| Gellus | 99.80 | 63.40 | +36.40 | ConNER |
| CLL | 95.70 | 85.98 | — | (Keine SOTA ausgestellt) |
| FSU | 96.10 | — | — | (Keine SOTA ausgestellt) |
† Closed-Source-Bewertungen werden von den besten veröffentlichten, von Experten begutachteten oder bewerteten Ergebnissen in der Literatur abgeleitet (in der Regel kommerzielle Modelle wie Spark NLP, NEEDLE, BERN2 usw.).
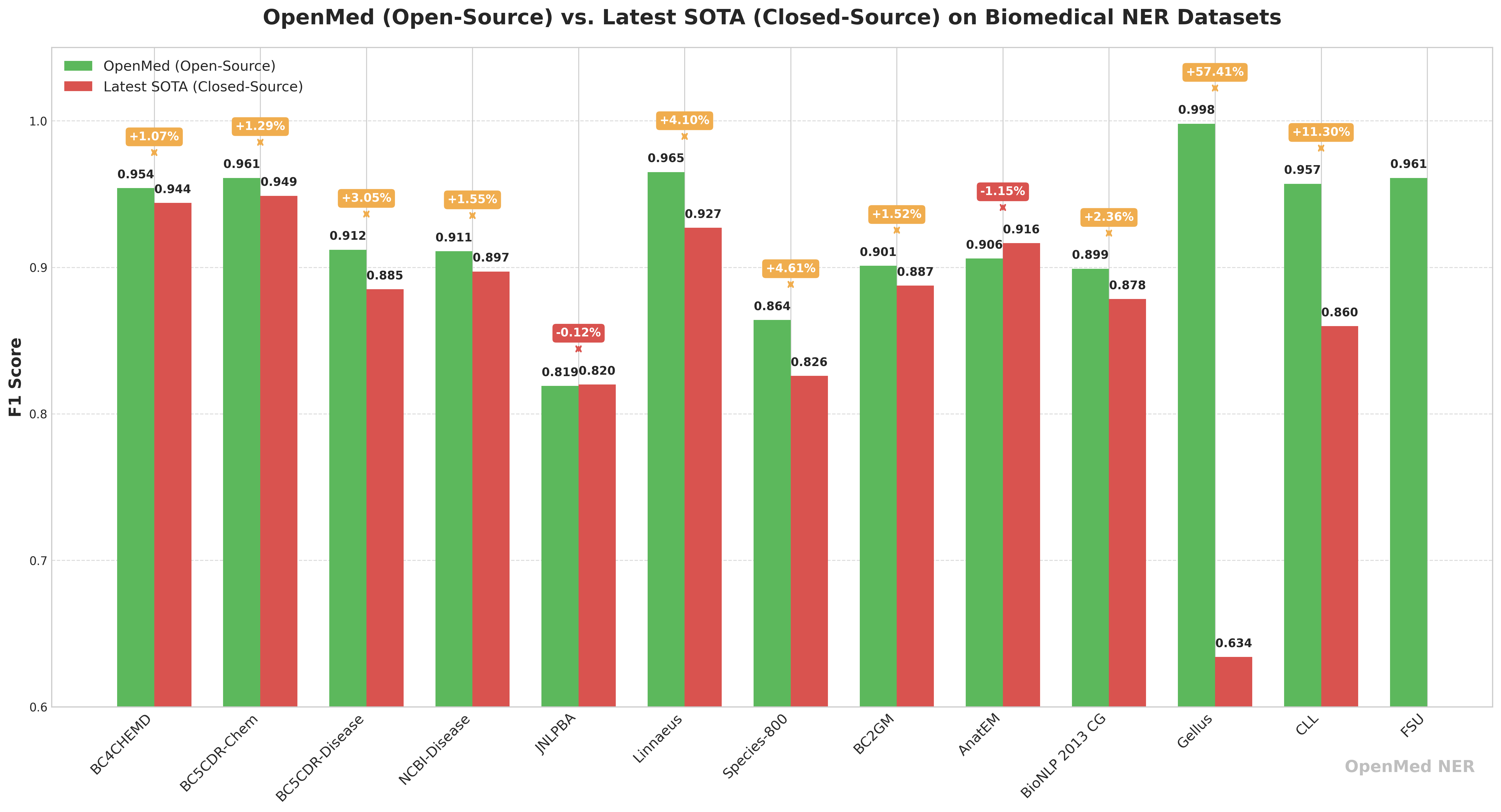
Besonders besorgniserregend ist die Tatsache, dass in Gellus auf den Datensatz.OpenMed Die F1-Punktzahl von 36,41 TP3T ist höher als die des vorhergehenden besten Modells, was darauf hindeutet, dass auf Optimierung ausgerichtete Open-Source-Modelle bei bestimmten Aufgaben ein großes Potenzial haben.
🔬 Nach Anwendungsbereich
Die folgende Tabelle ordnet die Datensätze den entsprechenden Bereichen des Gesundheitswesens zu und empfiehlt die geeigneten Modelle auf der Grundlage der kombinierten Leistung der Datensätze in jedem Bereich.
| Reich | Enthaltene Datensätze | Anzahl der verfügbaren Modelle | Bereich der Parameter | Empfehlungsmodelle |
|---|---|---|---|---|
| Pharmakologie | bc5cdr_chem, bc4chemd, fsu |
90 | 109M – 568M | OpenMed-NER-PharmaDetect-SuperClinical-434M |
| Krankheit/Pathologie | bc5cdr_disease, ncbi_disease |
60 | 109M – 434M | OpenMed-NER-PathologyDetect-PubMed-v2-109M |
| Genomik | jnlpba, bc2gm, species800, linnaeus, gellus |
150 | 335M – 568M | OpenMed-NER-GenomicDetect-SnowMed-568M |
| Anatomie | anatomy |
30 | 560M | OpenMed-NER-AnatomyDetect-ElectraMed-560M |
| Untersuchung von Tumoren | bionlp2013_cg |
30 | 355M | OpenMed-NER-OncologyDetect-SuperMedical-355M |
| Krankenakte | cll |
30 | 560M | OpenMed-NER-BloodCancerDetect-ElectraMed-560M |
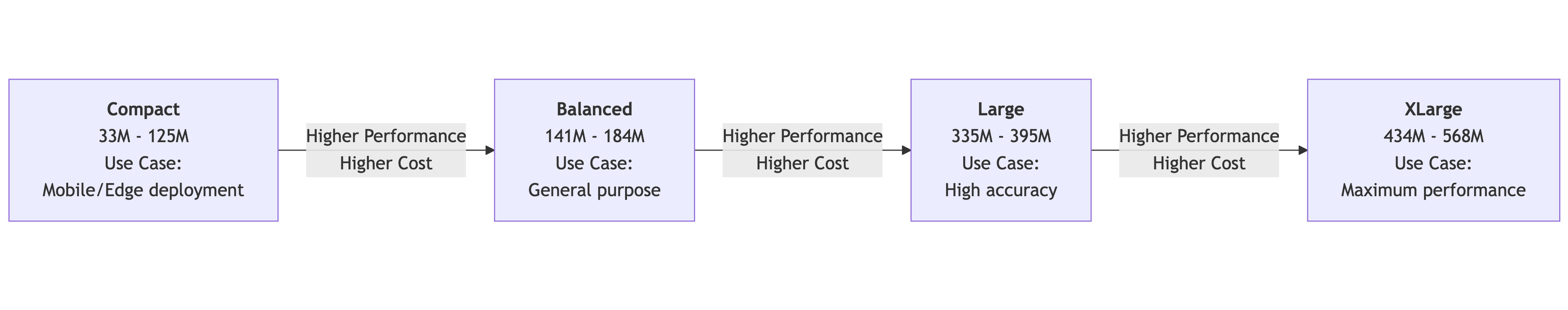
⚡️ Nach Modellgröße auswählen
| Größenordnung | Anzahl der Teilnehmer | Optimale Szenarien |
|---|---|---|
| kompakt | 109M | Schnelles Prototyping und ressourcenschonende Umgebung |
| Großes | 335M – 355M | Ausgewogene Wahl von Genauigkeit und Leistung |
| mega | 434M | Allrounder mit hervorragender Leistung |
| riesig | 560M – 568M | Aufgaben für höchste Präzision |
📊 Das beste Modell für jeden Datensatz
Nachfolgend finden Sie eine Zusammenfassung der Modelle mit den besten Ergebnissen für jeden Datensatz sowie deren F1-Werte und Größen.
| Datensatz | bestes Modell | F1 Ergebnis | Modellgröße (Parameter) |
|---|---|---|---|
bc5cdr_chem |
OpenMed-NER-PharmaDetect-SuperClinical-434M |
0.961 | 434M |
bionlp2013_cg |
OpenMed-NER-OncologyDetect-SuperMedical-355M |
0.899 | 355M |
bc4chemd |
OpenMed-NER-ChemicalDetect-PubMed-335M |
0.954 | 335M |
linnaeus |
OpenMed-NER-SpeciesDetect-PubMed-335M |
0.965 | 335M |
jnlpba |
OpenMed-NER-DNADetect-SuperClinical-434M |
0.819 | 434M |
bc5cdr_disease |
OpenMed-NER-DiseaseDetect-SuperClinical-434M |
0.912 | 434M |
fsu |
OpenMed-NER-ProteinDetect-SnowMed-568M |
0.961 | 568M |
ncbi_disease |
OpenMed-NER-PathologyDetect-PubMed-v2-109M |
0.911 | 109M |
bc2gm |
OpenMed-NER-GenomeDetect-SuperClinical-434M |
0.901 | 434M |
cll |
OpenMed-NER-BloodCancerDetect-ElectraMed-560M |
0.957 | 560M |
gellus |
OpenMed-NER-GenomicDetect-SnowMed-568M |
0.998 | 568M |
anatomy |
OpenMed-NER-AnatomyDetect-ElectraMed-560M |
0.906 | 560M |
species800 |
OpenMed-NER-OrganismDetect-BioMed-335M |
0.864 | 335M |
Schneller Einstieg und Skalierung
Hebelwirkung Hugging Face Transformers Bibliothek, Integration OpenMed Der Modellierungsprozess ist sehr einfach und kann in nur drei Codezeilen aufgerufen werden.
from transformers import pipeline
ner_pipeline = pipeline("token-classification", model="OpenMed/OpenMed-NER-PharmaDetect-SuperClinical-434M", aggregation_strategy="simple")
text = "Patient prescribed 10mg aspirin for hypertension."
entities = ner_pipeline(text)
print(entities)
# 输出: [{'entity_group': 'CHEMICAL', 'score': 0.99..., 'word': 'aspirin', 'start': 28, 'end': 35}]
Für Szenarien, in denen große Datenmengen verarbeitet werden müssen, bietet das Projekt auch effiziente Stapelverarbeitungslösungen.
from transformers.pipelines.pt_utils import KeyDataset
from datasets import Dataset, load_dataset
import pandas as pd
# 加载公开的医疗数据集(使用一个子集进行测试)
medical_dataset = load_dataset("BI55/MedText", split="train[:100]")
data = pd.DataFrame({"text": medical_dataset["Completion"]})
dataset = Dataset.from_pandas(data)
# 使用适合您硬件的批处理大小
batch_size = 16 # 根据您的 GPU 显存进行调整
results = []
ner_pipeline = pipeline("token-classification", model="OpenMed/OpenMed-NER-PharmaDetect-SuperClinical-434M", device=0) # 使用GPU
for out in ner_pipeline(KeyDataset(dataset, "text"), batch_size=batch_size):
results.extend(out)
print(f"批处理完成 {len(results)} 条文本")
NER Der reale Wert der freigeschalteten Technologie
Die Technologie der Erkennung benannter Entitäten (Named Entity Recognition, NER) ist in der Lage, automatisch Schlüsselinformationen aus unstrukturiertem Text zu extrahieren und zu klassifizieren. Im Gesundheitswesen ist diese Technologie ein Katalysator für die Aktivierung des Werts riesiger Datenmengen in klinischen Aufzeichnungen, Patientenakten und wissenschaftlicher Literatur.
- 🔒 Schutz der Privatsphäre der Patienten (De-Identifizierung von Daten):
NERPersönliche Gesundheitsinformationen (PHI) wie Name, Adresse usw. können automatisch identifiziert und aus Krankenakten entfernt werden. Dies ist wichtig für den Schutz der Privatsphäre der Patienten und die Einhaltung derHIPAAund anderen Gesetzen und Vorschriften und stellt gleichzeitig eine konforme und sichere Datenquelle für die medizinische Forschung dar, die weitaus effizienter und genauer ist als eine manuelle Verarbeitung. - 🔗 Konstruktion eines medizinischen Wissensgraphen (Extraktion von Entitätsbeziehungen)Nach der Identifizierung von Entitäten wie Medikamenten, Krankheiten usw. können weitere Techniken die Beziehungen zwischen ihnen analysieren (z. B. "Medikament A verursacht Nebenwirkung B"). Dies hilft beim Aufbau eines medizinischen Wissensgraphen, der die klinische Entscheidungsfindung unterstützt, die Arzneimittelentwicklung beschleunigt und letztlich eine personalisierte Behandlung ermöglicht.
- 💡 Optimierung der Kosten und des Managements im Gesundheitswesen (HCC-Codes)Hierarchical Condition Category (HCC) Kodierung ist ein wichtiges Verfahren, das von den Kostenträgern im Gesundheitswesen, wie Medicare, verwendet wird, um Kosten zu prognostizieren und Erstattungssätze festzulegen.
NERDiagnostische Informationen können automatisch aus medizinischen Aufzeichnungen extrahiert werden, um die Kodierung zu unterstützen. So wird sichergestellt, dass die Leistungserbringer für die Behandlung komplexer Fälle gerecht entlohnt werden, und gleichzeitig können Hochrisikopatienten für proaktive Maßnahmen identifiziert werden.
Durch die Förderung der Automatisierung dieser kritischen Aufgaben wird dieNER Die Technologie verwandelt schlummernde medizinische Texte in umsetzbare Lösungen, die die Datensicherheit erhöhen, die Forschung beschleunigen, die Patientenprognose verbessern und die Betriebskosten senken.OpenMed Das Entstehen eines neuen Systems wird diesen Prozess zweifellos erheblich beschleunigen.