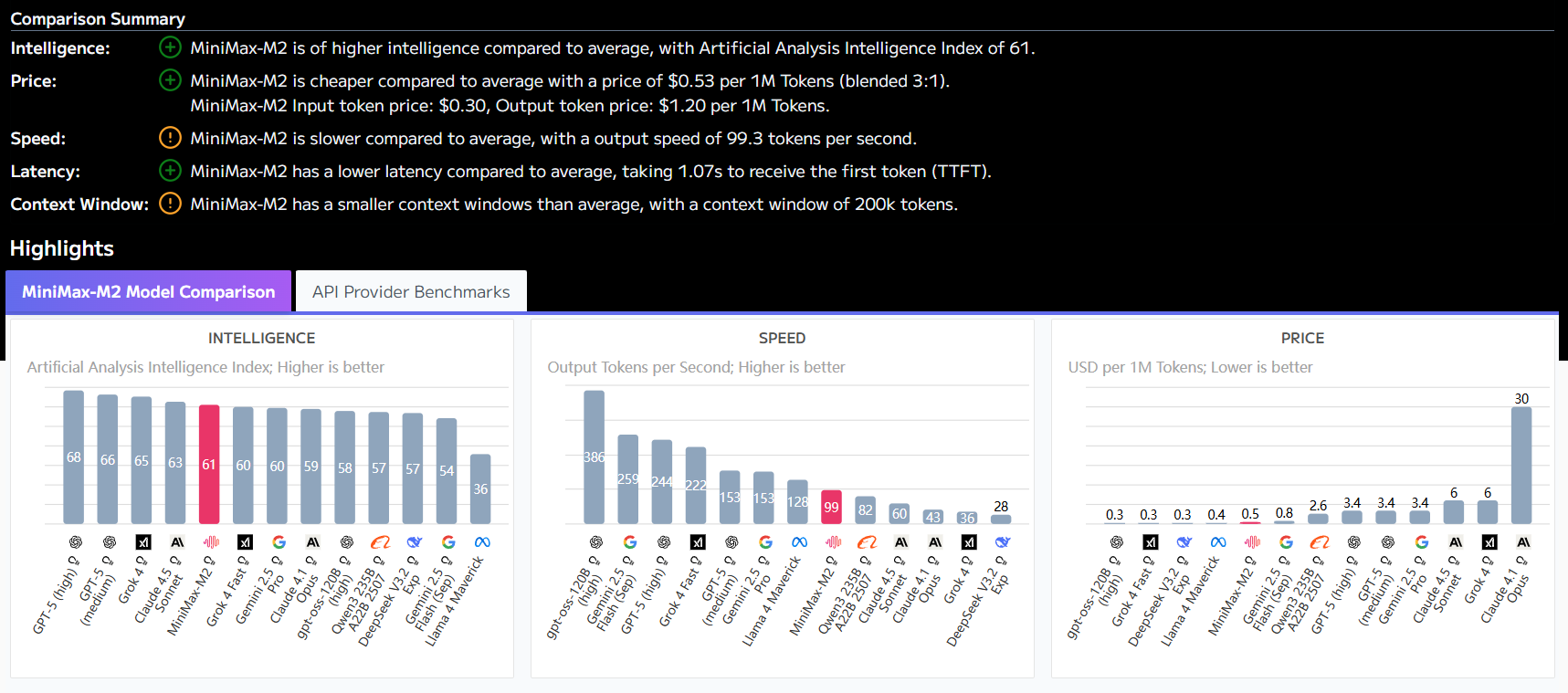
Kürzlich gaben OpenAI und das Biotech-Startup Retro Biosciences die Ergebnisse einer Zusammenarbeit bekannt, die das enorme Potenzial der künstlichen Intelligenz in den Biowissenschaften zeigt. Mit Hilfe eines KI-Modells, GPT-4b micro, das speziell für das Protein-Engineering entwickelt wurde, gelang es dem Team, die Expressionseffizienz von Schlüsselmarkern für die induzierte Reprogrammierung von Stammzellen um mehr als das 50-fache zu verbessern.
Im Mittelpunkt dieser Forschung stehen die Yamanaka-Faktoren, eine Gruppe von Proteinen, die für ihre bahnbrechende Rolle bei der Reprogrammierung von Zellen mit dem Nobelpreis ausgezeichnet wurden. Diese Proteine können differenzierte adulte Zellen, wie z. B. Hautzellen, in "induzierte pluripotente Stammzellen" (iPSC) verwandeln, die das Potenzial haben, sich zu fast jedem Gewebe zu entwickeln. Diese Fähigkeit eröffnet neue Wege zur Behandlung von Blindheit, Diabetes, Unfruchtbarkeit und sogar zur Behebung von Organmangel.
Die Anwendung herkömmlicher Yamanaka-Faktoren ist jedoch äußerst ineffizient: In der Regel werden weniger als 0,1% der Zellen erfolgreich transformiert, und der gesamte Prozess dauert mehr als drei Wochen. Die Effizienz sinkt noch weiter, insbesondere wenn es sich um Zellen von älteren oder kranken Spendern handelt. Diesmal hat die von der KI neu entwickelte Proteinvariante nicht nur die Effizienz deutlich verbessert, sondern auch eine größere Fähigkeit zur Reparatur von DNA-Schäden gezeigt, was bedeutet, dass sie ein größeres Potenzial zur Zellverjüngung hat.
Diese erste Entdeckung aus dem Jahr 2025 wurde nun durch wiederholte Experimente mit einer Vielzahl von Spendern, Zelltypen und Verabreichungsmethoden validiert, die die vollständige Pluripotenz und genomische Stabilität der resultierenden iPSC-Zelllinien bestätigen.
Experimentelle GPT-Modelle für das Protein-Engineering maßgeschneidert
Um zu beweisen, dass KI die biowissenschaftliche Forschung beschleunigen kann, hat OpenAI ein maßgeschneidertes Modell namens GPT-4b micro entwickelt. Das Modell ist eine Miniaturversion von GPT-4o, die speziell trainiert wurde, um ihr tiefes Wissen über Biologie zu vermitteln, insbesondere in den Bereichen Protein-Engineering, um Kontrollierbarkeit und Flexibilität zu gewährleisten.
Anders als die meisten Proteinsprachmodelle enthalten die Trainingsdaten für GPT-4b micro nicht nur Proteinsequenzen, sondern auch eine Mischung aus biologischem Text und markierten 3D-Strukturdaten. Die Trainingsdaten sind besonders reich an kontextuellen Informationen, wie z. B. Textbeschreibungen von Proteinen, homologe Sequenzen, die sich gemeinsam entwickelt haben, und bekannte interagierende Proteome. Dieser Ansatz ermöglicht es dem Modell, Sequenzen auf der Grundlage von Hinweisen aus spezifischen Attributen zu generieren und strukturierte Proteine und "intrinsisch ungeordnete" Proteine gleichermaßen gut zu behandeln. Bei den Yamanaka-Faktoren handelt es sich um letztere, deren Aktivität von einer großen Anzahl vorübergehender Wechselwirkungen mit mehreren Bindungspartnern und nicht von einer festen, stabilen Einzelstruktur abhängt.
Auf diese Weise übersteigt die effektive Kontextlänge des Modells die Grenze der unabhängigen Sequenzen bei weitem, und es können bis zu 64.000 Sequenzen im Inferenzprozess verarbeitet werden token Dies ist ein Novum bei der Modellierung von Proteinsequenzen.
Künstliche Intelligenz unterstützt die Transformation von SOX2 und KLF4
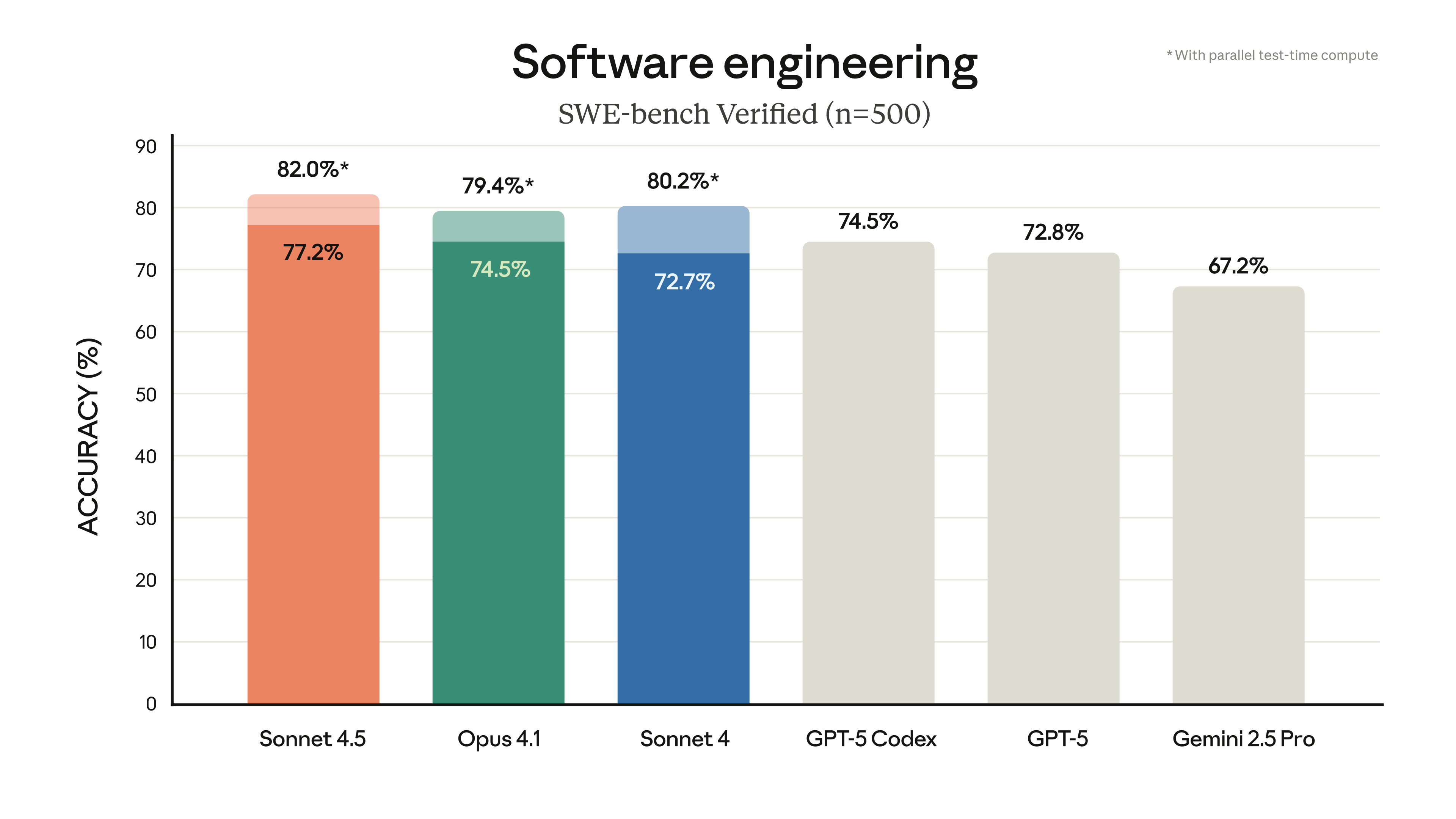
Der Yamanaka-Faktor besteht aus vier Proteinen: OCT4, SOX2, KLF4 und MYC (kurz: OSKM). Sie direkt durch Veränderung der Proteinsequenzen zu optimieren, ist eine schwierige Aufgabe. Im Fall von SOX2 (mit 317 Aminosäuren) und KLF4 (mit 513 Aminosäuren) beträgt die Anzahl der möglichen Varianten beispielsweise bis zu 10 hoch 1000.
Herkömmliche Methoden der "gerichteten Evolution", bei denen jeweils nur einige wenige Aminosäurereste verändert werden, können nur einen winzigen Bruchteil der Möglichkeiten erforschen. Im Gegensatz dazu kann die KI einen viel größeren Designraum erforschen. Das Team von Retro Biosciences baute zunächst eine Screening-Plattform im Nasslabor und verwendete dann das GPT-4b-Mikro, um eine Reihe von Kandidatensequenzen namens "RetroSOX" zu erzeugen.
Die Ergebnisse waren überraschend: Im Screening übertrafen modellvorgeschlagene Sequenzen mit mehr als 30% das Wildtyp-SOX2 bei der Expression wichtiger Pluripotenzmarker, obwohl sich ihre Aminosäuresequenzen im Durchschnitt um mehr als 100 unterscheiden. Im Gegensatz dazu lagen die Treffer bei konventionellen Screens in der Regel unter 10%.
Als nächstes nahm das Team KLF4 ins Visier. Das Modell generierte 14 "RetroKLF"-Varianten, die die beste Kombination im RetroSOX-Bildschirm übertrafen, mit einer Trefferquote nahe 50%.
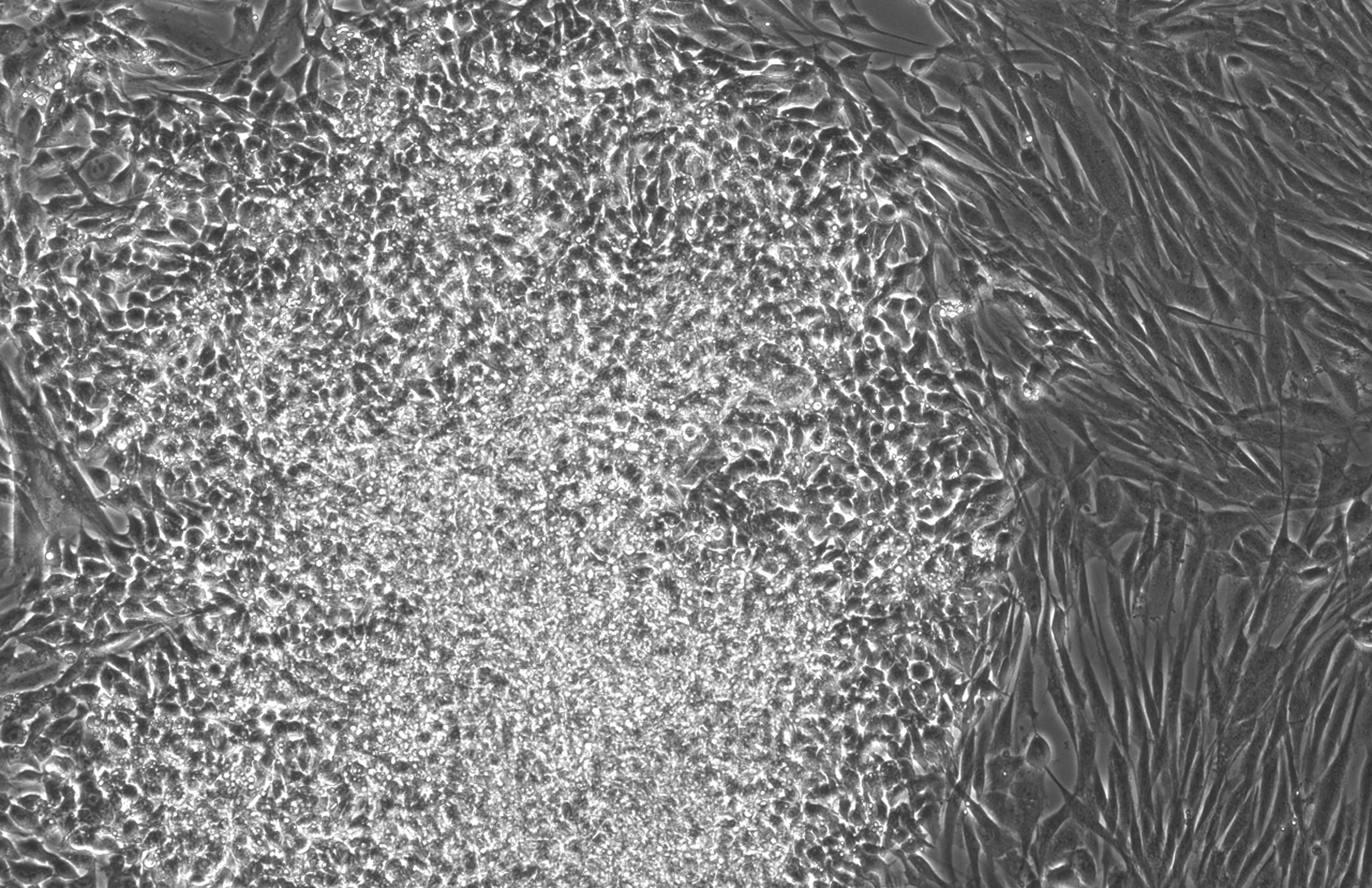
Die Auswirkungen waren am dramatischsten, wenn die Top-Varianten RetroSOX und RetroKLF kombiniert wurden. In drei unabhängigen Experimenten zeigten die Fibroblasten einen dramatischen Anstieg sowohl der frühen als auch der späten Pluripotenzmarker, und die späten Marker erschienen mehrere Tage früher als bei der Wildtyp-OSKM-Mischung. Weitere Tests, wie die Färbung mit alkalischer Phosphatase (AP), bestätigten ebenfalls, dass diese Zellkolonien nicht nur späte Marker exprimierten, sondern auch eine starke AP-Aktivität aufwiesen, ein starker Indikator für Pluripotenz.
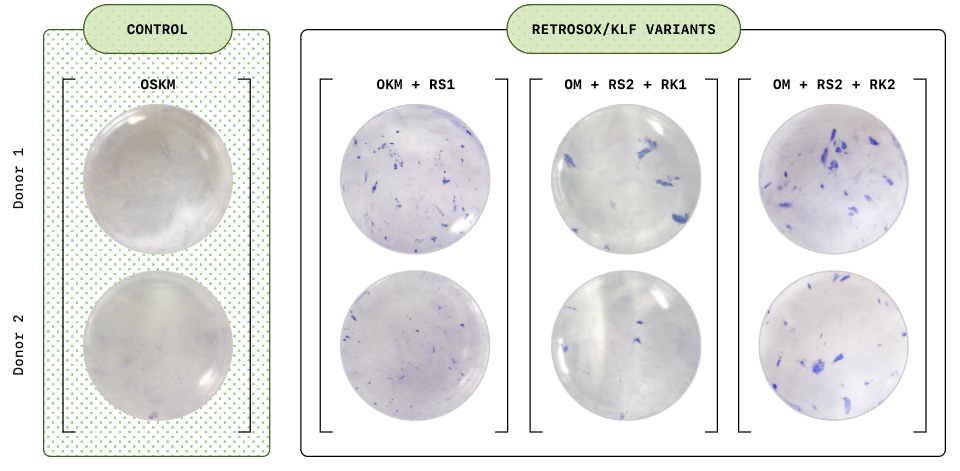
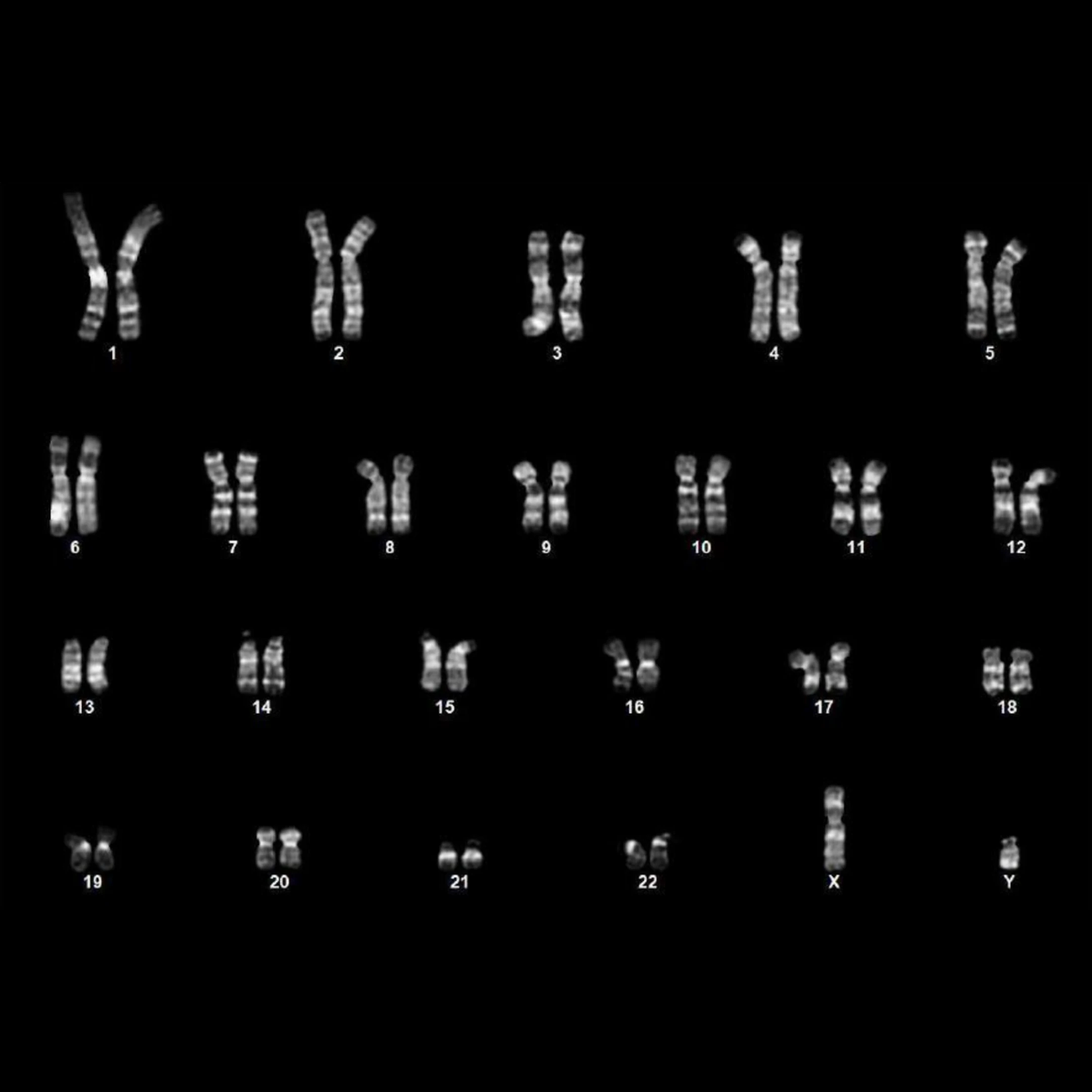
Um das klinische Potenzial zu erforschen, testete das Team auch eine andere Verabreichungsmethode (mRNA als Alternative zu viralen Vektoren) und einen anderen Zelltyp - mesenchymale Stromazellen (MSCs) von drei Spendern mittleren Alters über 50 Jahren. Innerhalb von nur 7 Tagen begannen mehr als 30%-Zellen, wichtige Pluripotenzmarker zu exprimieren; am 12. Tag aktivierten mehr als 85%-Zellen endogene Stammzellmarker wie OCT4 und NANOG. Die Karyotypisierung dieser Zellen zeigte eine normale Chromosomenstruktur, was ihre genomische Stabilität und ihre Eignung für die Zelltherapie bestätigte.
Verbesserte Reparatur von DNA-Schäden
Neben der Verbesserung der Reprogrammierungseffizienz untersuchten die Forscher das Potenzial dieser manipulierten Varianten für die zelluläre Verjüngung, insbesondere die Fähigkeit, DNA-Schäden zu reparieren, eines der klassischen Kennzeichen der zellulären Seneszenz.
Bei der Analyse von DNA-Schäden zeigten Zellen, die die RetroSOX/KLF-Mischung exprimierten, nach der Behandlung mit genotoxischen Chemikalien deutlich weniger DNA-Doppelstrangbruch-Marker (γ-H2AX-Signale) als Zellen, die Standard-OSKM oder Kontrollen verwendeten. Dies deutet darauf hin, dass die von AI entworfenen Proteinvarianten DNA-Schäden effizienter reparieren können, was einen neuen möglichen Weg zur Verzögerung der zellulären Seneszenz darstellt.

Zukunftsaussichten
Diese Arbeit zeigt deutlich, wie schnell ein domänenspezifisches KI-Modell einen Durchbruch bei gezielten wissenschaftlichen Problemen erzielen kann. Wenn Forscher tiefe Einblicke in das Fachgebiet mit Sprachmodellierungswerkzeugen kombinieren, können Probleme, deren Lösung früher Jahre dauerte, heute innerhalb von Tagen gelöst werden.
Natürlich befindet sich diese Forschung noch in einem frühen Stadium, und Sicherheit und langfristige Auswirkungen müssen noch sorgfältig bewertet werden, bevor sie vom Labor in die klinische Anwendung übergehen. Aber sie öffnet zweifellos eine neue Tür für die Anwendung von KI im Bereich der Biomedizin und läutet eine neue Ära der KI-gesteuerten personalisierten Medizin und der regenerativen Medizin ein.