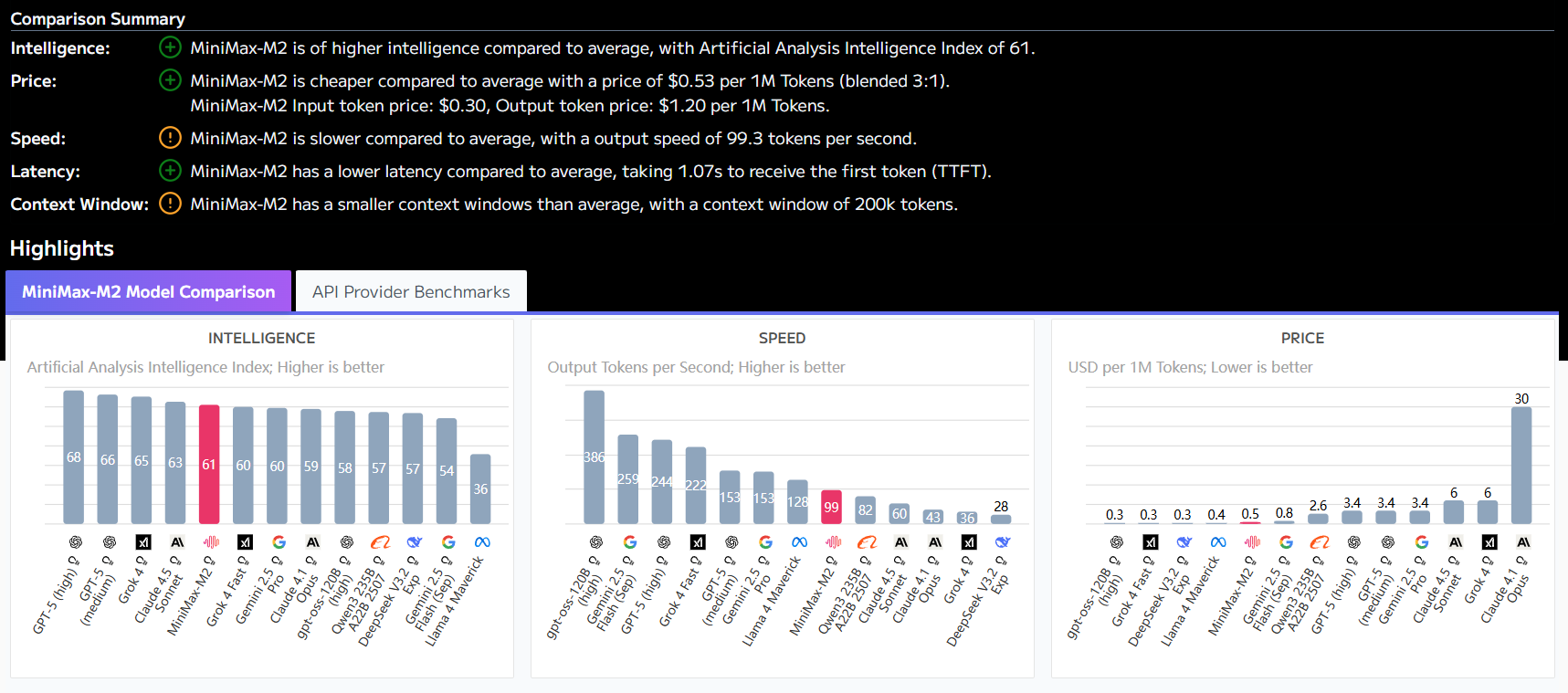
Evo 2のゼロサンプルのバリアント効果予測機能は、遺伝子バリアント効果を評価するための追加トレーニングを必要とせず、BRCA1遺伝子予測プロセスは以下の通りである:
予想ステップ
- データの準備参照配列とバリアント配列をリストに格納する
ref_seqs = ["ATCG..."] # 参考序列
var_seqs = ["ATGG..."] # 变体序列 - 業績予想::
ref_scores = model.score_sequences(ref_seqs)
var_scores = model.score_sequences(var_seqs)
print(f"Reference score: {ref_scores}, Variant score: {var_scores}")
結果の解釈
出力には参照配列とバリアント配列の尤度値が含まれます。両者の差を比較することで
- スコアが低いほど、そのバリアントは有害である可能性がある。
- わずかな変化は中立的な変種を示す
- 得点増加は機能獲得型変異体かもしれない
この機能は、特にがん関連遺伝子(BRCA1など)の病原性予測に応用でき、精密医療のための参照基盤を提供する。
この答えは記事から得たものである。Evo2: ゲノムモデリングとデザイン支援のためのオープンソースバイオAIツールについて