あまりにも長い間、最先端の医療AI技術は、高価な商用ライセンスと不透明な「ブラックボックス」システムの背後に閉じ込められてきた。このことが、多くの研究機関、中小規模の開発チーム、そして現場の医師たちの足かせとなり、技術革新と公平な導入のプロセスを遅らせてきた。そして今、次のような新しい取り組みが始まった。 OpenMed この行き詰まりを打破しようと、新しいプロジェクトが始動した。このプロジェクトは Hugging Face コミュニティは、380を超える先進的な医療および臨床テキストベースの名前付きエンティティ認識(NER)モデルをリリースし、それに基づき、以下のように発表した。 Apache 2.0 ライセンスは無料で、永続的に開放される。
この動きの意義は、無料の代替手段を提供したことだけでなく、公開したモデルが複数のベンチマークにおいて、主流の商用クローズドソース・ソリューションをも凌駕したという事実にもある。これは、ヘルスケアAIにおける技術的障壁が、オープンソースの力によって解体されつつあることを示している。
業界のジレンマとオープンソース・ソリューション
ヘルスケアAI分野は、成長を阻むいくつかの核となる障壁に直面している:
- 高額なライセンス料商用のトップAIツールのライセンスには高いコストがかかるため、予算が限られている学術機関や新興企業は敬遠される。
- 技術的な不透明度市販のツールは通常、そのモデルのアーキテクチャ、トレーニングデータ、動作を開示していないため、ユーザーがその信頼性や潜在的なバイアスを評価することは困難である。
- 技術革新の遅れ一部の有料モデルは、AI技術の最新の進歩に追いつくことができず、その性能は学界の最先端研究に徐々に遅れをとっている。
- 限定適用質の高いAIの能力は一部の大企業に集中しており、技術の普及を妨げている。
OpenMed このプロジェクトは、こうした課題に直接応えるものである。このプロジェクトは、380以上の無料の NER このモデルは、薬剤名、疾患、遺伝子、解剖学的構造など、医学テキスト中の主要な実体を識別することに重点を置いている。これらのモデルは以下のような特徴を持っている:
- ✅ 完全無料をベースにしている。
Apache 2.0自由な使用、改変、配布を許可するライセンス。 - ✅ すぐに使える実戦を想定して設計されているため、余計な手間をかけずに導入できる。
- ✅ サイズの柔軟性モデル・パラメーターの数は、異なるハードウェア要件に適合するように、109Mから568Mの範囲となっている。
- ✅ 実証済み医療分野における13以上の標準データセットで厳密な性能テストを実施。
- ✅ エコロジカル・コンパチブル:: と連携している。
Hugging Face和PyTorch以下のような主流のフレームワークとシームレスに統合できる。
OpenMed ツールキット詳細
OpenMed モデル・ライブラリは、慎重に微調整され、テストされた。 Gellus などのデータセットでのF1スコア 0.998その優れた性能を証明している。
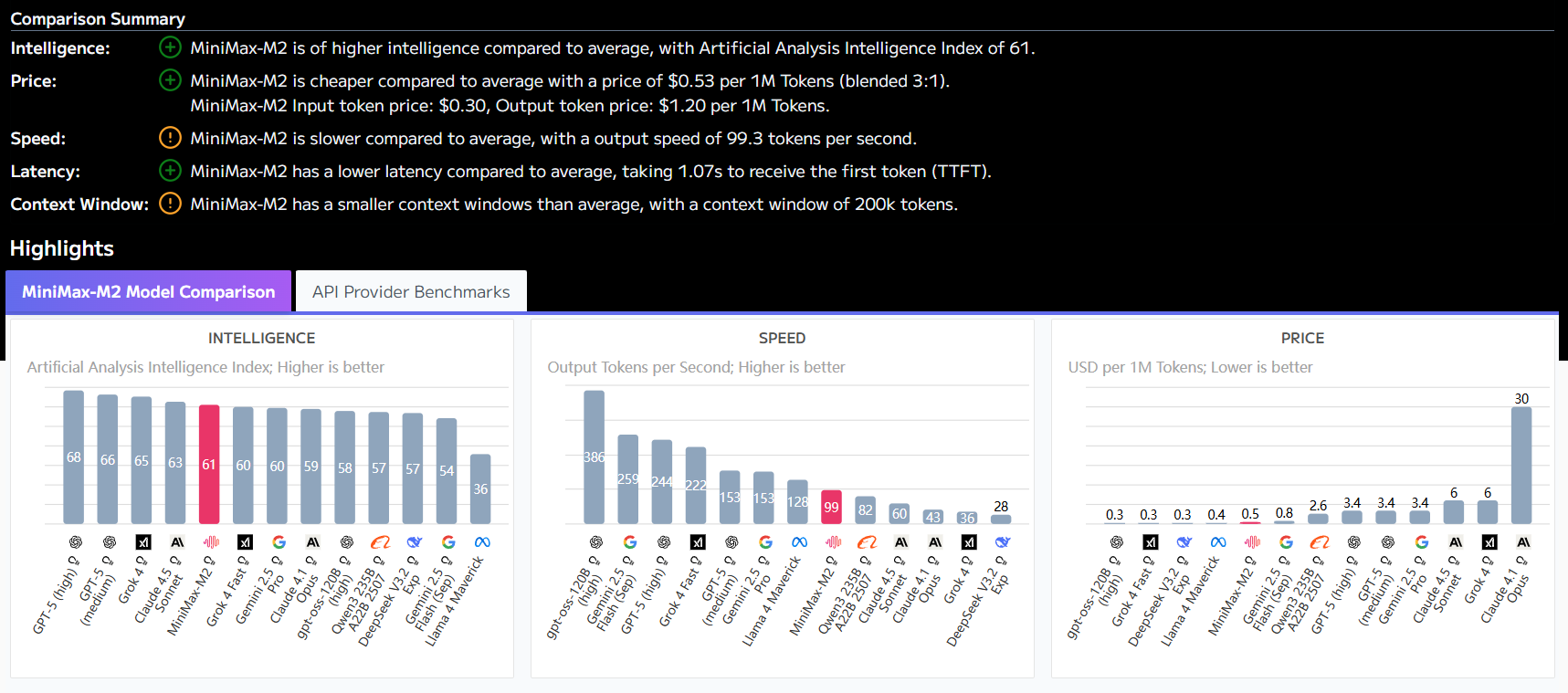
ᔬ 性能比較:オープンソース OpenMed クローズドソースのビジネスモデル
競争力を可視化するためにOpenMed は、現在の最先端のクローズドソースビジネスモデルとのパフォーマンス比較を発表した。そのデータによるとOpenMed いくつかのデータセットにおいて、市販のモデルに匹敵するだけでなく、いくつかのシナリオでは大幅な性能向上を達成している。
| データセット | オープンメッドベストF1スコア(%) | クローズド・ソース SOTA F1 フラクション(%†) | ギャップ(オープンメッド-SOTA) | 現在のクローズドソースのリーダー |
|---|---|---|---|---|
| BC4CHEMD | 95.40 | 94.39 | +1.01 | Spark NLP BertForTokenClassification |
| BC5CDR-Chem | 96.10 | 94.88 | +1.22 | Spark NLP BertForTokenClassification |
| BC5CDR-Disease | 91.20 | 88.5 | +2.70 | BioMegatron |
| NCBI-Disease | 91.10 | 89.71 | +1.39 | BioBERT |
| JNLPBA | 81.90 | 82.00 | –0.10 | KeBioLM (knowledge-enhanced LM) |
| Linnaeus | 96.50 | 92.70 | +3.80 | BERN2 toolkit |
| Species-800 | 86.40 | 82.59 | +3.81 | Spark NLP BertForTokenClassification |
| BC2GM | 90.10 | 88.75 | +1.35 | Spark NLP Bi-LSTM-CNN-Char |
| AnatEM | 90.60 | 91.65 | –1.05 | Spark NLP BertForTokenClassification |
| BioNLP 2013 CG | 89.90 | 87.83 | +2.07 | Spark NLP BertForTokenClassification |
| Gellus | 99.80 | 63.40 | +36.40 | ConNER |
| CLL | 95.70 | 85.98 | — | (SOTAは発行されない) |
| FSU | 96.10 | — | — | (SOTAは発行されない) |
クローズドソースのスコアは、文献の中で最も高く査読またはランク付けされた結果(通常、Spark NLP、NEEDLE、BERN2 などの商用モデル)から導き出される。
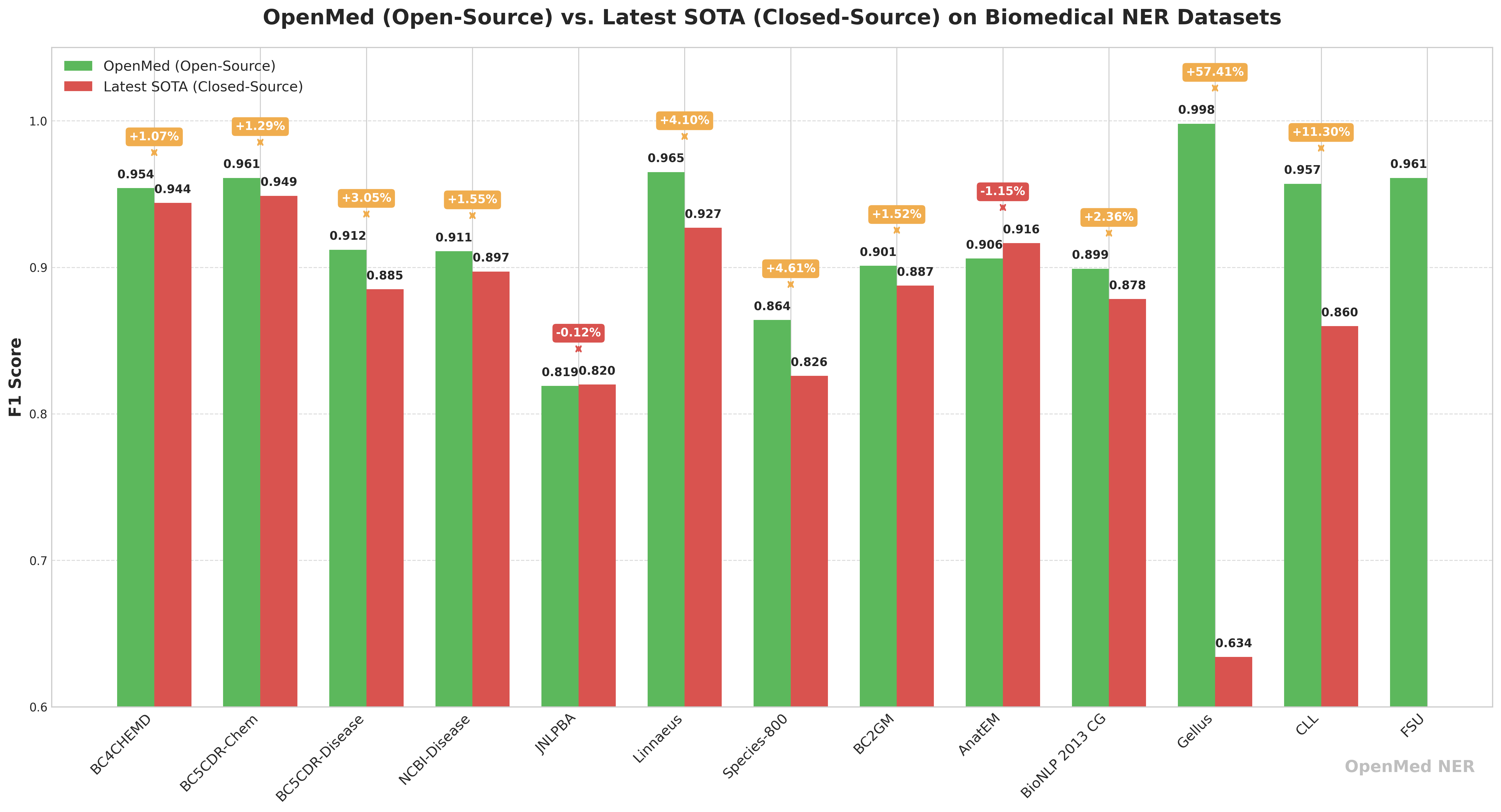
特に懸念されるのは Gellus データセット上で。OpenMed 36.4%のF1スコアは、以前のベストモデルよりも高く、最適化に焦点を当てたオープンソースモデルが、特定のタスクにおいて大きな可能性を秘めていることを示唆している。
ᔬ 応用分野別
次の表は、データセットを対応するヘルスケア領域にマッピングし、各領域におけるデータセットの総合的なパフォーマンスに基づいて適切なモデルを推奨している。
| レアル | 含まれるデータセット | モデル数 | パラメータの範囲 | 推薦モデル |
|---|---|---|---|---|
| 薬理学 | bc5cdr_chem, bc4chemd, fsu |
90 | 109M – 568M | OpenMed-NER-PharmaDetect-SuperClinical-434M |
| 疾患/病理学 | bc5cdr_disease, ncbi_disease |
60 | 109M – 434M | OpenMed-NER-PathologyDetect-PubMed-v2-109M |
| ゲノミクス | jnlpba, bc2gm, species800, linnaeus, gellus |
150 | 335M – 568M | OpenMed-NER-GenomicDetect-SnowMed-568M |
| 解剖学 | anatomy |
30 | 560M | OpenMed-NER-AnatomyDetect-ElectraMed-560M |
| 腫瘍研究 | bionlp2013_cg |
30 | 355M | OpenMed-NER-OncologyDetect-SuperMedical-355M |
| カルテ | cll |
30 | 560M | OpenMed-NER-BloodCancerDetect-ElectraMed-560M |
⚡️ サイズで選ぶ
| マグニチュード | 参加者数 | 最適なシナリオ |
|---|---|---|
| コンパクト | 109M | ラピッドプロトタイピングと低リソース環境 |
| メガ | 335M – 355M | 精度と性能のバランスの取れた選択 |
| メガ | 434M | 優れた性能を持つオールラウンダー |
| ジャイアント | 560M – 568M | 極めて精密な作業 |
📊 各データセットにおける最良のモデル
以下は、各データセットで最も優れたパフォーマンスを示したモデルと、そのF1スコアとサイズの要約である。
| データセット | ベストモデル | F1スコア | モデルサイズ(パラメータ) |
|---|---|---|---|
bc5cdr_chem |
OpenMed-NER-PharmaDetect-SuperClinical-434M |
0.961 | 434M |
bionlp2013_cg |
OpenMed-NER-OncologyDetect-SuperMedical-355M |
0.899 | 355M |
bc4chemd |
OpenMed-NER-ChemicalDetect-PubMed-335M |
0.954 | 335M |
linnaeus |
OpenMed-NER-SpeciesDetect-PubMed-335M |
0.965 | 335M |
jnlpba |
OpenMed-NER-DNADetect-SuperClinical-434M |
0.819 | 434M |
bc5cdr_disease |
OpenMed-NER-DiseaseDetect-SuperClinical-434M |
0.912 | 434M |
fsu |
OpenMed-NER-ProteinDetect-SnowMed-568M |
0.961 | 568M |
ncbi_disease |
OpenMed-NER-PathologyDetect-PubMed-v2-109M |
0.911 | 109M |
bc2gm |
OpenMed-NER-GenomeDetect-SuperClinical-434M |
0.901 | 434M |
cll |
OpenMed-NER-BloodCancerDetect-ElectraMed-560M |
0.957 | 560M |
gellus |
OpenMed-NER-GenomicDetect-SnowMed-568M |
0.998 | 568M |
anatomy |
OpenMed-NER-AnatomyDetect-ElectraMed-560M |
0.906 | 560M |
species800 |
OpenMed-NER-OrganismDetect-BioMed-335M |
0.864 | 335M |
迅速なスタートとスケーリング
レバレッジ Hugging Face Transformers ライブラリー, 統合 OpenMed モデリング・プロセスは非常にシンプルで、わずか3行のコードで呼び出すことができる。
from transformers import pipeline
ner_pipeline = pipeline("token-classification", model="OpenMed/OpenMed-NER-PharmaDetect-SuperClinical-434M", aggregation_strategy="simple")
text = "Patient prescribed 10mg aspirin for hypertension."
entities = ner_pipeline(text)
print(entities)
# 输出: [{'entity_group': 'CHEMICAL', 'score': 0.99..., 'word': 'aspirin', 'start': 28, 'end': 35}]
大規模なデータセットを処理する必要があるシナリオのために、プロジェクトは効率的なバッチ処理ソリューションも提供している。
from transformers.pipelines.pt_utils import KeyDataset
from datasets import Dataset, load_dataset
import pandas as pd
# 加载公开的医疗数据集(使用一个子集进行测试)
medical_dataset = load_dataset("BI55/MedText", split="train[:100]")
data = pd.DataFrame({"text": medical_dataset["Completion"]})
dataset = Dataset.from_pandas(data)
# 使用适合您硬件的批处理大小
batch_size = 16 # 根据您的 GPU 显存进行调整
results = []
ner_pipeline = pipeline("token-classification", model="OpenMed/OpenMed-NER-PharmaDetect-SuperClinical-434M", device=0) # 使用GPU
for out in ner_pipeline(KeyDataset(dataset, "text"), batch_size=batch_size):
results.extend(out)
print(f"批处理完成 {len(results)} 条文本")
NER ロック解除されたテクノロジーの実世界での価値
名前付き固有表現認識(NER)技術は、構造化されていないテキストから重要な情報を自動的に抽出し、分類することができる。ヘルスケアにおいて、この技術は、臨床記録、患者記録、科学文献に含まれる膨大なデータの価値を活性化する触媒となる。
- 🔒 患者のプライバシー保護(データの非識別化):
NER氏名、住所などの個人健康情報(PHI)は自動的に識別され、医療記録から削除することができる。これは、患者のプライバシーを保護し、以下を遵守する上で重要である。HIPAAまた、手作業で処理するよりも遥かに効率的で正確な、医療研究用のコンプライアンスに則った安全なデータソースを提供する。 - 医療知識グラフの構築(固有表現抽出)薬や病気などの実体を特定した後、さらなる技術によってそれらの間の関係(例えば「薬Aが副作用Bを引き起こす」)を分析することができる。これにより、臨床上の意思決定をサポートし、医薬品開発を加速させ、最終的には個別化治療を可能にする医療知識グラフを構築することができる。
- 💡 医療費と管理の最適化(HCCコード)HCC(Hierarchical Condition Category:階層的病態分類)コーディングは、メディケアなどの医療費支払者がコストを予測し、償還率を設定するために使用する重要なプロセスである。
NER医療記録から診断情報を自動的に抽出してコーディングに役立てることができるため、医療提供者は複雑な症例の治療に対して正当な報酬を得ることができる。
これらの重要な作業の自動化を推進することでNER テクノロジーは、眠っていた医学テキストを、データ・セキュリティの強化、研究の加速、患者の予後の改善、運用コストの削減を実現する実用的なソリューションへと変貌させつつある。OpenMed 新しいシステムの登場は、間違いなくこのプロセスを大幅に加速させるだろう。